本文简要介绍 python 语言中 scipy.stats.goodness_of_fit 的用法。
用法:
scipy.stats.goodness_of_fit(dist, data, *, known_params=None, fit_params=None, guessed_params=None, statistic='ad', n_mc_samples=9999, random_state=None)#执行拟合优度检验,将数据与分布族进行比较。
给定一个分布族和数据,对数据是从该族的分布中提取的原假设进行检验。可以指定任何已知的分布参数。分布的其余参数将适合数据,并相应地计算检验的 p 值。可以使用一些统计数据来比较分布与数据。
- dist: scipy.stats.rv_continuous
代表原假设下分布族的对象。
- data: 一维数组
有待测试的有限的、未经审查的数据。
- known_params: 字典,可选
包含 name-value 对已知分布参数的字典。蒙特卡罗样本是使用这些参数值从 null-hypothesized 分布中随机抽取的。在对每个蒙特卡洛样本进行统计评估之前,仅 null-hypothesized 分布族的剩余未知参数适合样本;已知参数保持固定。如果分布族的所有参数已知,则省略将分布族拟合到每个样本的步骤。
- fit_params: 字典,可选
包含已适合数据的name-value 分布参数对的字典,例如使用scipy.stats.fit或者
fit的方法距离。蒙特卡罗样本是使用这些指定的参数值从 null-hypothesized 分布中抽取的。然而,在这些蒙特卡罗样本上,null-hypothesized 分布族的这些参数和所有其他未知参数在评估统计数据之前已拟合。- guessed_params: 字典,可选
包含name-value对已被猜测的分布参数的字典。这些参数始终被视为自由参数,并且既适合所提供的数据,也适合从 null-hypothesized 分布中提取的蒙特卡洛样本。这些guessed_params的目的是用作数值拟合过程的初始值。
- statistic: {“ad”, “ks”, “cvm”, “filliben”},可选
将分布族的未知参数拟合到数据后,用于将数据与分布进行比较的统计量。 Anderson-Darling (“ad”) [1]、Kolmogorov-Smirnov (“ks”) [1]、Cramer-von Mises (“cvm”) [1] 和 Filliben (“filliben”) [7] 统计数据为可用的。
- n_mc_samples: 整数,默认值:9999
从零假设分布中抽取以形成统计零分布的蒙特卡罗样本数。每个样本的大小与给定数据相同。
- random_state: {无,整数,
numpy.random.Generator, numpy.random.RandomState}, optional用于生成蒙特卡罗样本的伪随机数生成器状态。
如果random_state是
None(默认),numpy.random.RandomState使用单例。如果random_state是一个 int,一个新的RandomState使用实例,播种random_state.如果random_state已经是一个Generator或者RandomState实例,然后使用提供的实例。
- res: GoodnessOfFitResult
具有以下属性的对象。
- fit_result
FitResult 表示所提供的距离与数据的拟合度的对象。该对象包括完全定义 null-hypothesized 分布(即从中抽取蒙特卡罗样本的分布)的分布族参数值。
- 统计 浮点数
将提供的数据与 null-hypothesized 分布进行比较的统计值。
- p值 浮点数
零分布中统计值至少与所提供数据的统计值一样极端的元素比例。
- null_distribution ndarray
从 null-hypothesized 分布中提取的每个蒙特卡洛样本的统计值。
- fit_result
参数 ::
返回 ::
注意:
这是一个广义的蒙特卡洛 goodness-of-fit 过程,其特殊情况对应于各种 Anderson-Darling 测试、Lilliefors 测试等。该测试在 [2]、[3] 和 [4] 中说明为参数引导程序测试。这是蒙特卡罗测试,其中指定样本分布的参数是根据数据估计的。我们自始至终都使用 “Monte Carlo” 而不是 “parametric bootstrap” 来说明测试,以避免与更熟悉的非参数引导程序混淆,并在下面说明如何执行测试。
传统的拟合优度检验
传统上,与一组固定的显著性水平相对应的临界值是使用蒙特卡罗方法预先计算的。用户通过仅计算其观察数据的检验统计量值并将该值与列表中的临界值进行比较来执行测试。这种做法不是很灵活,因为表格不适用于已知和未知参数值的所有分布和组合。此外,当从有限的表格数据中插入临界值以与用户的样本大小和拟合参数值相对应时,结果可能不准确。为了克服这些缺点,该函数允许用户执行适合其特定数据的蒙特卡罗试验。
算法概述
简而言之,该例程执行以下步骤:
Fit unknown parameters to the given data, thereby forming the “null-hypothesized” distribution, and compute the statistic of this pair of data and distribution.
Draw random samples from this null-hypothesized distribution.
Fit the unknown parameters to each random sample.
Calculate the statistic between each sample and the distribution that has been fit to the sample.
Compare the value of the statistic corresponding with data from (1) against the values of the statistic corresponding with the random samples from (4). The p-value is the proportion of samples with a statistic value greater than or equal to the statistic of the observed data.
更详细地,步骤如下。
首先,由指定的分布族的任何未知参数距离适合所提供的数据使用最大似然估计。 (一个例外是位置和尺度未知的正态分布:我们使用偏差校正标准差
np.std(data, ddof=1)对于建议的规模[1].) 这些参数值指定称为“null-hypothesized 分布”的分布族的特定成员,即在原假设下对数据进行采样的分布。这统计,将数据与分布进行比较,计算结果为数据和null-hypothesized分布。接下来,从 null-hypothesized 分布中抽取许多(具体为 n_mc_samples)新样本,每个样本包含与数据相同数量的观察值。分布族 dist 的所有未知参数都适合每个重采样,并且在每个样本与其相应的拟合分布之间计算统计量。这些统计值形成了蒙特卡洛零分布(不要与上面的“null-hypothesized 分布”混淆)。
检验的 p 值是蒙特卡罗零分布中至少与所提供数据的统计值一样极端的统计值的比例。更准确地说,p 值由下式给出
其中是蒙特卡罗零分布中大于或等于计算的统计值的统计值的数量数据, 和是蒙特卡罗零分布中的元素数量 (n_mc_samples)。添加的分子和分母可以被认为包括与数据在零分布中,但更正式的解释在[5].
限制
对于某些分布族,测试可能非常慢,因为分布族的未知参数必须适合每个蒙特卡罗样本,并且对于 SciPy 中的大多数分布,通过数值优化执行分布拟合。
反模式
因此,将(由用户)预先拟合数据的分布参数视为 known_params 可能很诱人,因为分布的所有参数的规范排除了将分布拟合到每个 Monte 的需要。卡洛样本。 (这本质上就是原始 Kilmogorov-Smirnov 检验的执行方式。)尽管这样的检验可以提供反对零假设的证据,但该检验是保守的,因为较小的 p 值往往会(大大)高估使I 类错误(即,尽管原假设为真,但仍拒绝原假设),并且检验的功效较低(即,即使原假设为假,也不太可能拒绝原假设)。这是因为蒙特卡罗样本不太可能与 null-hypothesized 分布以及数据一致。这往往会增加零分布中记录的统计值,从而使更多的统计值超过数据的统计值,从而夸大 p 值。
参考:
[1] (1,2,3,4,5)M.A.斯蒂芬斯(1974)。 “EDF 拟合优度统计数据和一些比较。”美国统计协会杂志,卷。 69,第 730-737 页。
[2]W. Stute、W. G. Manteiga 和 M. P. Quindimil (1993)。 “基于引导goodness-of-fit-tests。”梅特里卡 40.1:243-256。
[3]C. Genest 和 B Rémillard。 (2008)。 “半参数模型中 goodness-of-fit 测试的参数引导程序的有效性。” 《IHP 概率与统计年鉴》。卷。 44.第6号。
[4]I.Kojadinovic 和 J.Yan (2012)。 “Goodness-of-fit 基于加权引导程序的测试:参数引导程序的快速 large-sample 替代方案。”加拿大统计杂志 40.3:480-500。
[5]B. Phipson 和 G. K. Smyth (2010)。 “排列 P 值不应该为零:随机抽取排列时计算精确的 P 值。” 9.1 遗传学和分子生物学中的统计应用
[6]H.W.利利福斯 (1967)。 “关于均值和方差未知的 Kolmogorov-Smirnov 正态性检验。”美国统计协会杂志 62.318:399-402。
[7]Filliben,James J.“正态性的概率图相关系数检验。”技术计量学 17.1 (1975):111-117。
例子:
数据从给定分布中抽取的原假设的一个众所周知的检验是 Kolmogorov-Smirnov (KS) 检验,在 SciPy 中以
scipy.stats.ks_1samp形式提供。假设我们希望测试以下数据是否:>>> import numpy as np >>> from scipy import stats >>> rng = np.random.default_rng() >>> x = stats.uniform.rvs(size=75, random_state=rng)从正态分布中抽样。为了执行 KS 检验,观测数据的经验分布函数将与正态分布的(理论)累积分布函数进行比较。当然,为此,必须完全指定原假设下的正态分布。这通常是通过首先将分布的
loc和scale参数拟合到观察到的数据,然后执行测试来完成的。>>> loc, scale = np.mean(x), np.std(x, ddof=1) >>> cdf = stats.norm(loc, scale).cdf >>> stats.ks_1samp(x, cdf) KstestResult(statistic=0.1119257570456813, pvalue=0.2827756409939257)KS-test 的优点是可以准确、高效地计算 p 值(在零假设下获得与从观测数据获得的值一样极端的检验统计量值的概率)。
goodness_of_fit只能近似这些结果。>>> known_params = {'loc': loc, 'scale': scale} >>> res = stats.goodness_of_fit(stats.norm, x, known_params=known_params, ... statistic='ks', random_state=rng) >>> res.statistic, res.pvalue (0.1119257570456813, 0.2788)统计数据完全匹配,但 p 值是通过形成“蒙特卡洛零分布”来估计的,即通过使用提供的参数从
scipy.stats.norm中显式抽取随机样本并计算每个样本的统计量。这些统计值的分数至少与res.statistic一样极端,近似于scipy.stats.ks_1samp计算的精确 p 值。然而,在许多情况下,我们更愿意只测试数据是从以下之一采样的:任何正态分布家族的成员,不是专门来自位置和尺度适合观察样本的正态分布。在这种情况下,利利福斯[6]认为 KS 检验过于保守(即 p 值高估了拒绝真实原假设的实际概率),因此缺乏功效——当原假设实际上为假时拒绝原假设的能力。事实上,我们上面的 p 值约为 0.28,这个值太大,无法在任何常见的显著性水平上拒绝原假设。
考虑一下为什么会这样。请注意,在上面的 KS 检验中,统计量始终将数据与拟合到的正态分布的 CDF 进行比较观测数据。这往往会降低观测数据的统计值,但在计算其他样本的统计值时,例如我们随机抽取形成蒙特卡罗零分布的样本,它就是“unfair”。纠正这一点很容易:每当我们计算样本的 KS 统计量时,我们都会使用拟合的正态分布的 CDF那个样本。这种情况下的零分布尚未被精确计算,并且通常使用如上所述的蒙特卡罗方法来近似。这是哪里
goodness_of_fit表现出色。>>> res = stats.goodness_of_fit(stats.norm, x, statistic='ks', ... random_state=rng) >>> res.statistic, res.pvalue (0.1119257570456813, 0.0196)事实上,这个 p 值要小得多,并且小到足以(正确地)拒绝常见显著性水平(包括 5% 和 2.5%)的原假设。
然而,KS 统计量对所有偏离正态性的情况都不是很敏感。 KS 统计量的最初优点是能够从理论上计算零分布,但现在可以使用更敏感的统计量(导致更高的测试功效),因为我们可以通过计算来近似零分布。 Anderson-Darling 统计量 [1] 往往更加敏感,并且已使用蒙特卡罗方法针对各种显著性水平和样本量对该统计量的临界值进行了制表。
>>> res = stats.anderson(x, 'norm') >>> print(res.statistic) 1.2139573337497467 >>> print(res.critical_values) [0.549 0.625 0.75 0.875 1.041] >>> print(res.significance_level) [15. 10. 5. 2.5 1. ]这里,统计量的观察值超过了对应于1%显著性水平的临界值。这告诉我们观测数据的 p 值小于 1%,但它是什么呢?我们可以根据这些 (already-interpolated) 值进行插值,但
goodness_of_fit可以直接估计它。>>> res = stats.goodness_of_fit(stats.norm, x, statistic='ad', ... random_state=rng) >>> res.statistic, res.pvalue (1.2139573337497467, 0.0034)另一个优点是,
goodness_of_fit的使用不限于参数已知与必须根据数据估计的一组特定分布或条件。相反,goodness_of_fit可以使用足够快速且可靠的fit方法相对快速地估计任何分布的 p 值。例如,在这里,我们使用 Cramer-von Mises 统计量针对已知位置和未知尺度的瑞利分布执行拟合优度检验。>>> rng = np.random.default_rng() >>> x = stats.chi(df=2.2, loc=0, scale=2).rvs(size=1000, random_state=rng) >>> res = stats.goodness_of_fit(stats.rayleigh, x, statistic='cvm', ... known_params={'loc': 0}, random_state=rng)执行速度相当快,但为了检查
fit方法的可靠性,我们应该检查拟合结果。>>> res.fit_result # location is as specified, and scale is reasonable params: FitParams(loc=0.0, scale=2.1026719844231243) success: True message: 'The fit was performed successfully.' >>> import matplotlib.pyplot as plt # matplotlib must be installed to plot >>> res.fit_result.plot() >>> plt.show()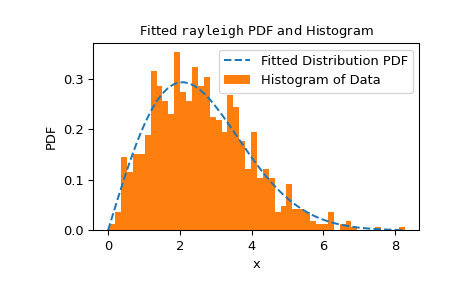
如果分布不太适合观察到的数据,则检验可能无法控制 I 类错误率,即拒绝原假设(即使原假设为真)的机会。
我们还应该寻找零分布中可能由不可靠拟合引起的极端异常值。这些并不一定会使结果无效,但它们往往会降低测试的功效。
>>> _, ax = plt.subplots() >>> ax.hist(np.log10(res.null_distribution)) >>> ax.set_xlabel("log10 of CVM statistic under the null hypothesis") >>> ax.set_ylabel("Frequency") >>> ax.set_title("Histogram of the Monte Carlo null distribution") >>> plt.show()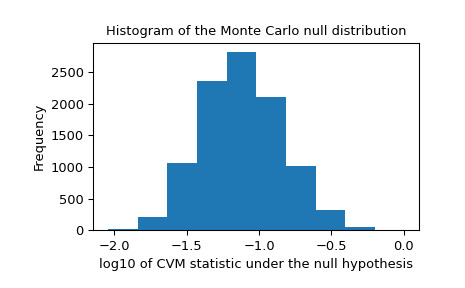
这个剧情看起来让人安心。
如果
fit方法工作可靠,并且检验统计量的分布对拟合参数的值不是特别敏感,则goodness_of_fit提供的 p 值预计将是一个很好的近似值。>>> res.statistic, res.pvalue (0.2231991510248692, 0.0525)
相关用法
- Python SciPy stats.gompertz用法及代码示例
- Python SciPy stats.genpareto用法及代码示例
- Python SciPy stats.gumbel_l用法及代码示例
- Python SciPy stats.gzscore用法及代码示例
- Python SciPy stats.genlogistic用法及代码示例
- Python SciPy stats.gennorm用法及代码示例
- Python SciPy stats.gibrat用法及代码示例
- Python SciPy stats.genhalflogistic用法及代码示例
- Python SciPy stats.gmean用法及代码示例
- Python SciPy stats.gamma用法及代码示例
- Python scipy.stats.gilbrat用法及代码示例
- Python SciPy stats.geom用法及代码示例
- Python SciPy stats.genexpon用法及代码示例
- Python SciPy stats.gstd用法及代码示例
- Python SciPy stats.genhyperbolic用法及代码示例
- Python SciPy stats.gaussian_kde用法及代码示例
- Python SciPy stats.gumbel_r用法及代码示例
- Python SciPy stats.gausshyper用法及代码示例
- Python SciPy stats.gengamma用法及代码示例
- Python SciPy stats.genextreme用法及代码示例
- Python SciPy stats.geninvgauss用法及代码示例
- Python SciPy stats.anderson用法及代码示例
- Python SciPy stats.iqr用法及代码示例
- Python SciPy stats.skewnorm用法及代码示例
- Python SciPy stats.cosine用法及代码示例
注:本文由纯净天空筛选整理自scipy.org大神的英文原创作品 scipy.stats.goodness_of_fit。非经特殊声明,原始代码版权归原作者所有,本译文未经允许或授权,请勿转载或复制。
