本文簡要介紹 python 語言中 scipy.stats.skewtest 的用法。
用法:
scipy.stats.skewtest(a, axis=0, nan_policy='propagate', alternative='two-sided')#測試偏態是否不同於正態分布。
此函數檢驗零假設,即抽取樣本的總體偏度與相應正態分布的偏度相同。
- a: 數組
要測試的數據。
- axis: int 或無,可選
計算統計數據的軸。默認值為 0。如果沒有,則計算整個數組 a。
- nan_policy: {‘propagate’, ‘raise’, ‘omit’},可選
定義當輸入包含 nan 時如何處理。以下選項可用(默認為‘propagate’):
‘propagate’:返回 nan
‘raise’:引發錯誤
‘omit’:執行忽略 nan 值的計算
- alternative: {‘雙麵’,‘less’, ‘greater’},可選
定義備擇假設。默認為“雙麵”。可以使用以下選項:
“雙麵”:樣本分布的偏度與正態分布的偏度不同(即 0)
‘less’:樣本底層分布的偏度小於正態分布的偏度
‘greater’:樣本底層分布的偏度大於正態分布的偏度
- statistic: 浮點數
此測試的計算 z-score。
- pvalue: 浮點數
假設檢驗的 p 值。
參數 ::
返回 ::
注意:
樣本量必須至少為 8。
參考:
[1]R. B. D'Agostino、A. J. Belanger 和 R. B. D'Agostino Jr.,“使用強大且信息豐富的正態性檢驗的建議”,美國統計學家 44,第 316-321 頁,1990。
[2]夏皮羅,S.S. 和威爾克,M.B. (1965)。正態性方差檢驗分析(完整樣本)。生物計量學,52(3/4),591-611。
[3]B. Phipson 和 G. K. Smyth。 “排列 P 值不應該為零:隨機抽取排列時計算精確的 P 值。”遺傳學和分子生物學中的統計應用 9.1 (2010)。
例子:
假設我們希望從測量中推斷醫學研究中成年男性的體重是否不服從正態分布 [2]。重量(磅)記錄在下麵的數組
x中。>>> import numpy as np >>> x = np.array([148, 154, 158, 160, 161, 162, 166, 170, 182, 195, 236])[1] 中的偏度測試首先根據樣本偏度計算統計量。
>>> from scipy import stats >>> res = stats.skewtest(x) >>> res.statistic 2.7788579769903414由於正態分布的偏度為零,因此對於從正態分布中抽取的樣本,該統計量的大小往往較低。
該檢驗是通過將統計量的觀測值與零分布進行比較來執行的:零分布是在權重從正態分布中得出的零假設下得出的統計值的分布。
對於此檢驗,非常大樣本的統計量的零分布是標準正態分布。
>>> import matplotlib.pyplot as plt >>> dist = stats.norm() >>> st_val = np.linspace(-5, 5, 100) >>> pdf = dist.pdf(st_val) >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> def st_plot(ax): # we'll re-use this ... ax.plot(st_val, pdf) ... ax.set_title("Skew Test Null Distribution") ... ax.set_xlabel("statistic") ... ax.set_ylabel("probability density") >>> st_plot(ax) >>> plt.show()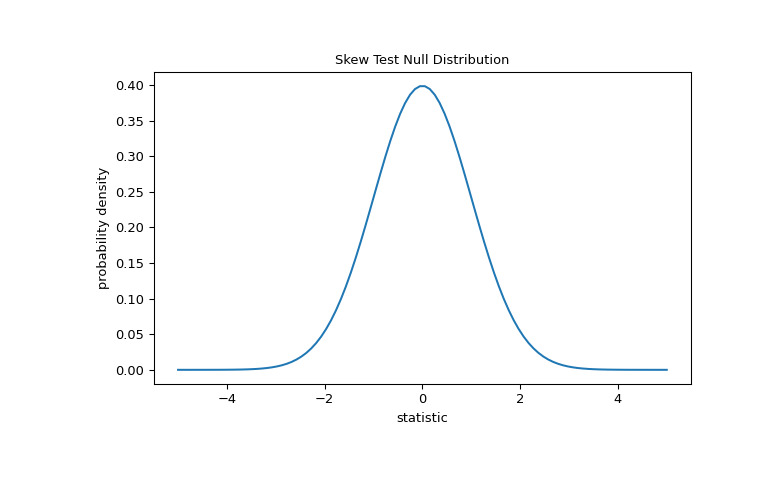
比較通過 p 值進行量化:零分布中比統計觀察值極端或更極端的值的比例。在雙邊測試中,大於觀察統計量的零分布元素和小於觀察統計量負值的零分布元素均被視為 “more extreme”。
>>> fig, ax = plt.subplots(figsize=(8, 5)) >>> st_plot(ax) >>> pvalue = dist.cdf(-res.statistic) + dist.sf(res.statistic) >>> annotation = (f'p-value={pvalue:.3f}\n(shaded area)') >>> props = dict(facecolor='black', width=1, headwidth=5, headlength=8) >>> _ = ax.annotate(annotation, (3, 0.005), (3.25, 0.02), arrowprops=props) >>> i = st_val >= res.statistic >>> ax.fill_between(st_val[i], y1=0, y2=pdf[i], color='C0') >>> i = st_val <= -res.statistic >>> ax.fill_between(st_val[i], y1=0, y2=pdf[i], color='C0') >>> ax.set_xlim(-5, 5) >>> ax.set_ylim(0, 0.1) >>> plt.show()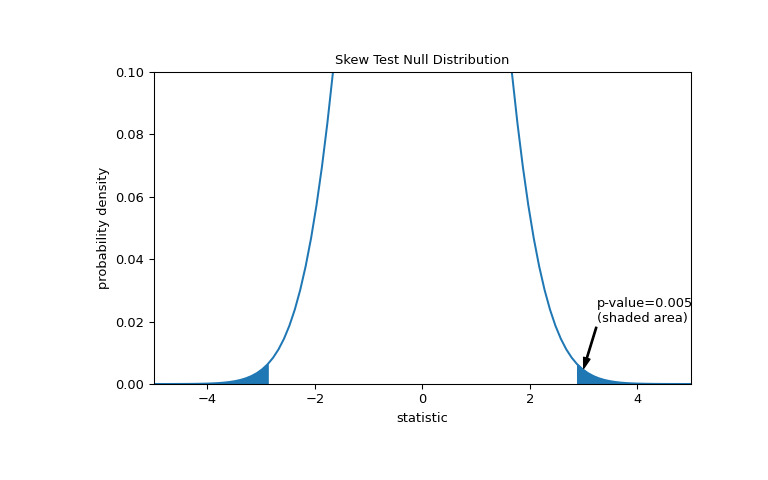
>>> res.pvalue 0.005455036974740185如果 p 值為 “small” - 也就是說,如果從正態分布總體中采樣數據產生統計數據的極值的概率較低 - 這可以作為反對零假設的證據另一種選擇:權重不是從正態分布中得出的。注意:
反之則不成立;也就是說,檢驗不用於為原假設提供證據。
將被視為 “small” 的值的閾值是在分析數據之前應做出的選擇 [3],同時考慮誤報(錯誤地拒絕原假設)和漏報(未能拒絕假設)的風險。錯誤的原假設)。
請注意,標準正態分布提供了零分布的漸近近似;它僅對於具有許多觀測值的樣本是準確的。對於像我們這樣的小樣本,
scipy.stats.monte_carlo_test可以提供更準確的(盡管是隨機的)精確 p 值的近似值。>>> def statistic(x, axis): ... # get just the skewtest statistic; ignore the p-value ... return stats.skewtest(x, axis=axis).statistic >>> res = stats.monte_carlo_test(x, stats.norm.rvs, statistic) >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> st_plot(ax) >>> ax.hist(res.null_distribution, np.linspace(-5, 5, 50), ... density=True) >>> ax.legend(['aymptotic approximation\n(many observations)', ... 'Monte Carlo approximation\n(11 observations)']) >>> plt.show()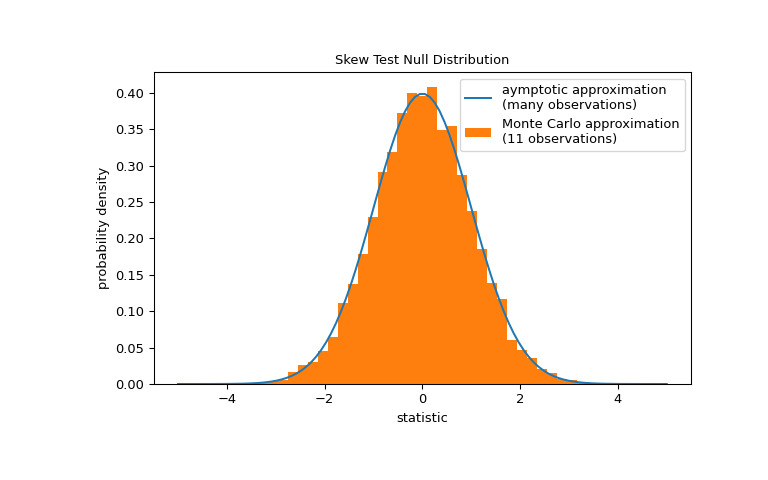
>>> res.pvalue 0.0062 # may vary在這種情況下,即使對於我們的小樣本,漸近近似和蒙特卡洛近似也相當接近。
相關用法
- Python SciPy stats.skewnorm用法及代碼示例
- Python SciPy stats.skewcauchy用法及代碼示例
- Python SciPy stats.skew用法及代碼示例
- Python SciPy stats.skellam用法及代碼示例
- Python SciPy stats.semicircular用法及代碼示例
- Python scipy.stats.somersd用法及代碼示例
- Python SciPy stats.sem用法及代碼示例
- Python SciPy stats.siegelslopes用法及代碼示例
- Python SciPy stats.scoreatpercentile用法及代碼示例
- Python SciPy stats.spearmanr用法及代碼示例
- Python SciPy stats.shapiro用法及代碼示例
- Python SciPy stats.studentized_range用法及代碼示例
- Python SciPy stats.sigmaclip用法及代碼示例
- Python SciPy stats.special_ortho_group用法及代碼示例
- Python SciPy stats.sobol_indices用法及代碼示例
- Python SciPy stats.anderson用法及代碼示例
- Python SciPy stats.iqr用法及代碼示例
- Python SciPy stats.genpareto用法及代碼示例
- Python SciPy stats.cosine用法及代碼示例
- Python SciPy stats.norminvgauss用法及代碼示例
- Python SciPy stats.directional_stats用法及代碼示例
- Python SciPy stats.invwishart用法及代碼示例
- Python SciPy stats.bartlett用法及代碼示例
- Python SciPy stats.levy_stable用法及代碼示例
- Python SciPy stats.page_trend_test用法及代碼示例
注:本文由純淨天空篩選整理自scipy.org大神的英文原創作品 scipy.stats.skewtest。非經特殊聲明,原始代碼版權歸原作者所有,本譯文未經允許或授權,請勿轉載或複製。
