本文简要介绍 python 语言中 scipy.stats.logrank 的用法。
用法:
scipy.stats.logrank(x, y, alternative='two-sided')#通过对数秩检验比较两个样本的生存分布。
- x, y: 数组 或 CensoredData
根据经验生存函数进行比较的样本。
- alternative: {‘双面’,‘less’, ‘greater’},可选
定义备择假设。
原假设是两个组(例如 X 和 Y)的生存分布是相同的。
以下替代假设 [4] 可用(默认为“双面”):
“双面”:两组的生存分布不相同。
‘less’:X组的生存受到青睐:X组的故障率函数有时小于Y组的故障率函数。
‘greater’:Y组的生存受到青睐:X组的故障率函数有时大于Y组的故障率函数。
- res: LogRankResult
包含属性的对象:
- 统计 浮点数数组
计算的统计量(定义如下)。它的大小是大多数其他对数秩测试实现返回的大小的平方根。
- p值 浮点数数组
测试的计算 p 值。
参数 ::
返回 ::
注意:
对数秩检验 [1] 在两个样本来自同一分布的原假设下,将观察到的事件数与预期事件数进行比较。统计数据是
其中
表示组(即,它可以采用值 或 ,或者可以省略引用组合样本) 表示时间(事件发生的时间), 是事件发生前处于危险中的受试者数量, 是当时观察到的事件数量。
logrank返回的statistic是许多其他实现返回的统计信息的(带符号)平方根。在零假设下, 根据一个自由度的卡方分布渐近分布。因此, 根据标准正态分布渐近分布。使用 的优点是保留了符号信息(即观察到的事件数量是否倾向于小于或大于原假设下的预期数量),从而允许scipy.stats.logrank提供单方面的替代假设。参考:
[1]Mantel N.“生存数据的评估和考虑中出现的两个新的排名顺序统计数据。”癌症化疗报告,50(3):163-170,PMID:5910392,1966
[2]布兰德·奥特曼,“The logrank test”,BMJ,328:1073,DOI:10.1136/bmj.328.7447.1073,2004 年
[3]“Logrank test”,维基百科,https://en.wikipedia.org/wiki/Logrank_test
[4]布朗、马克. “关于对数秩检验的方差选择。”生物计量学 71.1 (1984):65-74。
[5]约翰·P·克莱因 (Klein) 和梅尔文·L·莫施伯格 (Melvin L. Moeschberger)。生存分析:审查和截断数据的技术。卷。 1230.纽约:施普林格,2003年。
例子:
参考文献[2]比较了两种不同类型的复发性恶性胶质瘤患者的生存时间。下面的样本记录了每位患者参与研究的时间(周数)。使用
scipy.stats.CensoredData类是因为数据是right-censored:未经审查的观察结果与观察到的死亡相对应,而审查的观察结果与因其他原因离开研究的患者相对应。>>> from scipy import stats >>> x = stats.CensoredData( ... uncensored=[6, 13, 21, 30, 37, 38, 49, 50, ... 63, 79, 86, 98, 202, 219], ... right=[31, 47, 80, 82, 82, 149] ... ) >>> y = stats.CensoredData( ... uncensored=[10, 10, 12, 13, 14, 15, 16, 17, 18, 20, 24, 24, ... 25, 28,30, 33, 35, 37, 40, 40, 46, 48, 76, 81, ... 82, 91, 112, 181], ... right=[34, 40, 70] ... )我们可以计算并可视化两组的经验生存函数,如下所示。
>>> import numpy as np >>> import matplotlib.pyplot as plt >>> ax = plt.subplot() >>> ecdf_x = stats.ecdf(x) >>> ecdf_x.sf.plot(ax, label='Astrocytoma') >>> ecdf_y = stats.ecdf(y) >>> ecdf_x.sf.plot(ax, label='Glioblastoma') >>> ax.set_xlabel('Time to death (weeks)') >>> ax.set_ylabel('Empirical SF') >>> plt.legend() >>> plt.show()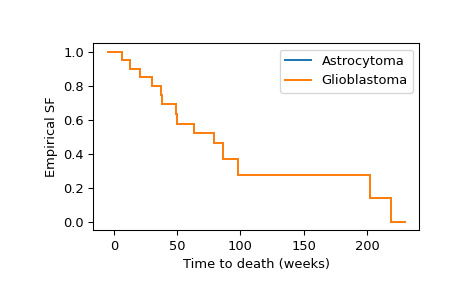
对经验生存函数的目视检查表明,两组之间的生存时间往往不同。为了正式评估差异在 1% 水平上是否显著,我们使用对数秩检验。
>>> res = stats.logrank(x=x, y=y) >>> res.statistic -2.73799... >>> res.pvalue 0.00618...p 值小于 1%,因此我们可以将这些数据视为反对原假设的证据,支持两个生存函数之间存在差异的替代方案。
相关用法
- Python SciPy stats.lognorm用法及代码示例
- Python SciPy stats.loglaplace用法及代码示例
- Python SciPy stats.loguniform用法及代码示例
- Python SciPy stats.logistic用法及代码示例
- Python SciPy stats.logser用法及代码示例
- Python SciPy stats.loggamma用法及代码示例
- Python SciPy stats.lomax用法及代码示例
- Python SciPy stats.levy_stable用法及代码示例
- Python SciPy stats.laplace用法及代码示例
- Python SciPy stats.levy_l用法及代码示例
- Python SciPy stats.linregress用法及代码示例
- Python SciPy stats.levene用法及代码示例
- Python SciPy stats.levy用法及代码示例
- Python SciPy stats.laplace_asymmetric用法及代码示例
- Python SciPy stats.anderson用法及代码示例
- Python SciPy stats.iqr用法及代码示例
- Python SciPy stats.genpareto用法及代码示例
- Python SciPy stats.skewnorm用法及代码示例
- Python SciPy stats.cosine用法及代码示例
- Python SciPy stats.norminvgauss用法及代码示例
- Python SciPy stats.directional_stats用法及代码示例
- Python SciPy stats.invwishart用法及代码示例
- Python SciPy stats.bartlett用法及代码示例
- Python SciPy stats.page_trend_test用法及代码示例
- Python SciPy stats.itemfreq用法及代码示例
注:本文由纯净天空筛选整理自scipy.org大神的英文原创作品 scipy.stats.logrank。非经特殊声明,原始代码版权归原作者所有,本译文未经允许或授权,请勿转载或复制。
