本文簡要介紹 python 語言中 scipy.stats.kendalltau 的用法。
用法:
scipy.stats.kendalltau(x, y, *, initial_lexsort=<object object>, nan_policy='propagate', method='auto', variant='b', alternative='two-sided')#計算 Kendall 的 tau,這是一種序數數據的相關性度量。
Kendall tau 是衡量兩個排名之間對應關係的指標。接近 1 的值表示強烈同意,接近 -1 的值表示強烈反對。這實現了 Kendall tau 的兩個變體:tau-b(默認)和 tau-c(也稱為 Stuart 的 tau-c)。它們的區別僅在於如何將它們歸一化到 -1 到 1 的範圍內;假設檢驗(它們的 p 值)是相同的。 Kendall 最初的tau-a 沒有單獨實現,因為tau-b 和tau-c 在沒有聯係的情況下都減少為tau-a。
- x, y: array_like
排列的數組,形狀相同。如果數組不是一維的,它們將被展平為一維。
- initial_lexsort: 布爾值,可選,已棄用
該參數未使用。
- nan_policy: {‘propagate’, ‘raise’, ‘omit’},可選
定義當輸入包含 nan 時如何處理。以下選項可用(默認為‘propagate’):
‘propagate’: returns nan
‘raise’: throws an error
‘omit’: performs the calculations ignoring nan values
- method: {‘auto’, ‘asymptotic’, ‘exact’},可選
定義使用哪種方法來計算 p 值 [5]。以下選項可用(默認為‘auto’):
‘auto’: selects the appropriate method based on a trade-off between speed and accuracy
‘asymptotic’: uses a normal approximation valid for large samples
‘exact’: computes the exact p-value, but can only be used if no ties are present. As the sample size increases, the ‘exact’ computation time may grow and the result may lose some precision.
- variant: {‘b’, ‘c’},可選
定義返回 Kendall tau 的哪個變體。默認為‘b’。
- alternative: {‘雙麵’,‘less’, ‘greater’},可選
定義備擇假設。默認為“雙麵”。可以使用以下選項:
“雙邊”:等級相關性非零
‘less’:秩相關為負(小於零)
‘greater’:秩相關為正(大於零)
- res: SignificanceResult
包含屬性的對象:
- 統計 浮點數
tau 統計量。
- p值 浮點數
假設檢驗的 p 值,其原假設是不存在關聯,tau = 0。
參數 ::
返回 ::
注意:
使用的 Kendall tau 定義為 [2]:
tau_b = (P - Q) / sqrt((P + Q + T) * (P + Q + U)) tau_c = 2 (P - Q) / (n**2 * (m - 1) / m)其中 P 是一致對的數量,Q 是不一致對的數量,T 是僅在 x 中的關係數,而 U 是僅在 y 中的關係數。如果同一對在 x 和 y 中出現平局,則不會將其添加到 T 或 U。n 是樣本總數,m 是 x 或 y 中唯一值的數量,以較小者為準。
參考:
[1]Maurice G. Kendall,“等級相關性的新度量”,Biometrika Vol。 30,第 1/2 期,第 81-93 頁,1938 年。
[2]Maurice G. Kendall,“排名問題中關係的處理”,Biometrika Vol。 33,第 3 期,第 239-251 頁。 1945 年。
[3]Gottfried E. Noether,“非參數統計要素”,John Wiley & Sons,1967 年。
[4]Peter M. Fenwick,“累積頻率表的新數據結構”,軟件:實踐與經驗,卷。 24,第 3 期,第 327-336 頁,1994 年。
[5]Maurice G. Kendall,“Rank Correlation Methods”(第 4 版),Charles Griffin & Co.,1970 年。
[6]Kershenobich, D.、Fierro, F. J. 和 Rojkind, M. (1970)。人肝硬化中遊離脯氨酸池與膠原蛋白含量的關係。臨床研究雜誌,49(12), 2246-2249。
[7]Hollander, M.、Wolfe, D. A. 和 Chicken, E. (2013)。非參數統計方法。約翰·威利父子。
[8]B. Phipson 和 G. K. Smyth。 “排列 P 值不應該為零:隨機抽取排列時計算精確的 P 值。”遺傳學和分子生物學中的統計應用 9.1 (2010)。
例子:
考慮以下來自[6]的數據,該數據研究了不健康人類肝髒中遊離脯氨酸(一種氨基酸)和總膠原蛋白(一種常見於結締組織中的蛋白質)之間的關係。
下麵的
x和y數組記錄了兩種化合物的測量值。觀察結果是配對的:每個遊離脯氨酸測量值均取自同一肝髒,作為相同index下的總膠原蛋白測量值。>>> import numpy as np >>> # total collagen (mg/g dry weight of liver) >>> x = np.array([7.1, 7.1, 7.2, 8.3, 9.4, 10.5, 11.4]) >>> # free proline (μ mole/g dry weight of liver) >>> y = np.array([2.8, 2.9, 2.8, 2.6, 3.5, 4.6, 5.0])[7] 使用 Spearman 相關係數對這些數據進行了分析,該統計量與 Kendall tau 類似,因為它對樣本之間的序數相關性也很敏感。讓我們使用 Kendall 的 tau 蛋白進行類似的研究。
>>> from scipy import stats >>> res = stats.kendalltau(x, y) >>> res.statistic 0.5499999999999999對於具有強正序相關的樣本,該統計量的值往往較高(接近 1);對於具有強負序相關的樣本,該統計量的值往往較低(接近 -1);對於具有強負序相關的樣本,該統計量的值往往較小(接近於 0)。具有弱序數相關性。
該檢驗是通過將統計觀察值與零分布進行比較來進行的:零假設下得出的統計值分布,即總膠原蛋白和遊離脯氨酸測量值是獨立的。
對於此測試,沒有聯係的大樣本的零分布近似為具有方差
(2*(2*n + 5))/(9*n*(n - 1))的正態分布,其中n = len(x)。>>> import matplotlib.pyplot as plt >>> n = len(x) # len(x) == len(y) >>> var = (2*(2*n + 5))/(9*n*(n - 1)) >>> dist = stats.norm(scale=np.sqrt(var)) >>> z_vals = np.linspace(-1.25, 1.25, 100) >>> pdf = dist.pdf(z_vals) >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> def plot(ax): # we'll re-use this ... ax.plot(z_vals, pdf) ... ax.set_title("Kendall Tau Test Null Distribution") ... ax.set_xlabel("statistic") ... ax.set_ylabel("probability density") >>> plot(ax) >>> plt.show()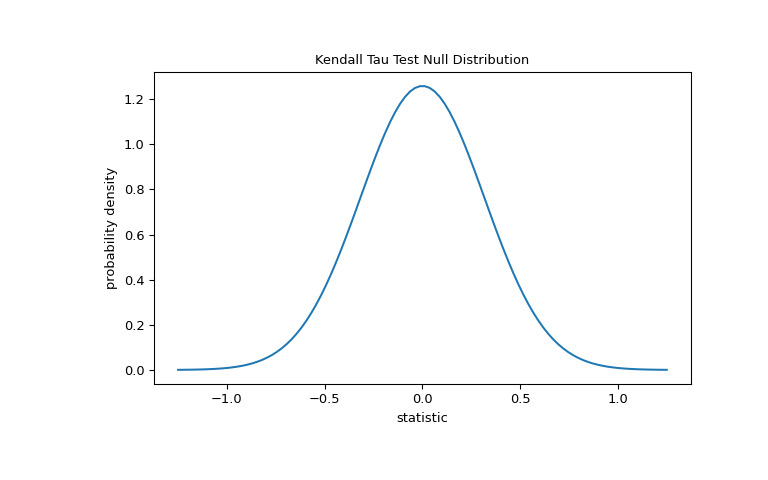
比較通過 p 值進行量化:零分布中比統計觀察值極端或更極端的值的比例。在統計量為正的雙邊測試中,大於轉換統計量的零分布元素和小於觀察統計量負值的零分布元素均被視為 “more extreme”。
>>> fig, ax = plt.subplots(figsize=(8, 5)) >>> plot(ax) >>> pvalue = dist.cdf(-res.statistic) + dist.sf(res.statistic) >>> annotation = (f'p-value={pvalue:.4f}\n(shaded area)') >>> props = dict(facecolor='black', width=1, headwidth=5, headlength=8) >>> _ = ax.annotate(annotation, (0.65, 0.15), (0.8, 0.3), arrowprops=props) >>> i = z_vals >= res.statistic >>> ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0') >>> i = z_vals <= -res.statistic >>> ax.fill_between(z_vals[i], y1=0, y2=pdf[i], color='C0') >>> ax.set_xlim(-1.25, 1.25) >>> ax.set_ylim(0, 0.5) >>> plt.show()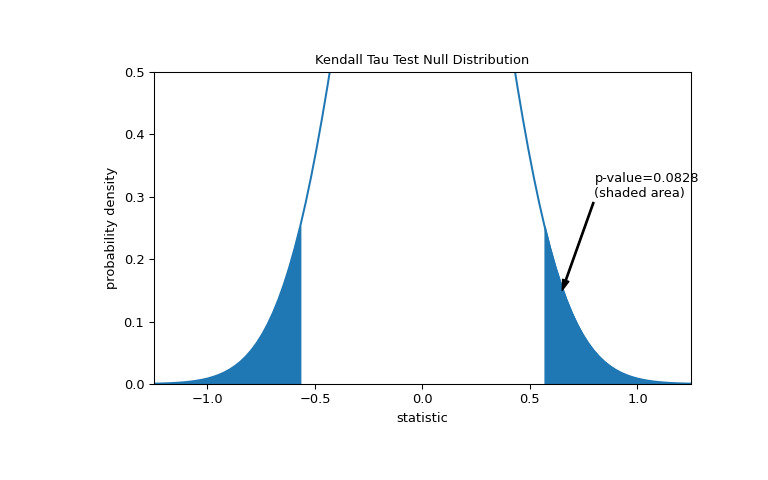
>>> res.pvalue 0.09108705741631495 # approximate p-value請注意,曲線的陰影區域與
kendalltau返回的 p 值之間存在輕微差異。這是因為我們的數據具有聯係,並且我們忽略了kendalltau執行的對零分布方差的聯係校正。對於沒有聯係的樣本,繪圖的陰影區域和kendalltau返回的 p 值將完全匹配。如果 p 值為 “small” - 也就是說,如果從產生如此統計量極值的獨立分布中采樣數據的概率較低 - 這可以作為反對零假設的證據,支持替代方案:總膠原蛋白和遊離脯氨酸的分布不是獨立的。注意:
反之則不成立;也就是說,檢驗不用於為原假設提供證據。
將被視為 “small” 的值的閾值是在分析數據之前應做出的選擇 [8],同時考慮誤報(錯誤地拒絕零假設)和漏報(未能拒絕假設)的風險。錯誤的原假設)。
p 值小並不能證明效果大;相反,它們隻能為 “significant” 效應提供證據,這意味著它們不太可能在原假設下發生。
對於沒有中等尺寸連接的樣品,
kendalltau可以準確計算 p 值。然而,在有聯係的情況下,kendalltau訴諸漸近近似。盡管如此,我們可以使用排列檢驗來準確計算零分布:在總膠原蛋白和遊離脯氨酸是獨立的零假設下,每個遊離脯氨酸測量值與任何總膠原蛋白測量值都有相同的可能被觀察到。因此,我們可以組建一個精確的通過計算之間每個可能的元素配對下的統計量來計算空分布x和y.>>> def statistic(x): # explore all possible pairings by permuting `x` ... return stats.kendalltau(x, y).statistic # ignore pvalue >>> ref = stats.permutation_test((x,), statistic, ... permutation_type='pairings') >>> fig, ax = plt.subplots(figsize=(8, 5)) >>> plot(ax) >>> bins = np.linspace(-1.25, 1.25, 25) >>> ax.hist(ref.null_distribution, bins=bins, density=True) >>> ax.legend(['aymptotic approximation\n(many observations)', ... 'exact null distribution']) >>> plot(ax) >>> plt.show()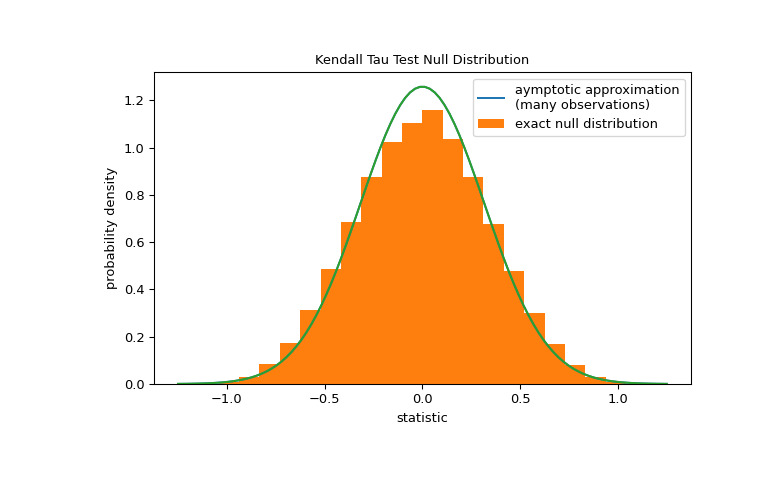
>>> ref.pvalue 0.12222222222222222 # exact p-value請注意,此處計算的精確 p 值與上麵
kendalltau返回的近似值之間存在顯著差異。對於有聯係的小樣本,請考慮執行排列檢驗以獲得更準確的結果。
相關用法
- Python SciPy stats.kurtosis用法及代碼示例
- Python SciPy stats.kruskal用法及代碼示例
- Python SciPy stats.kurtosistest用法及代碼示例
- Python SciPy stats.kappa3用法及代碼示例
- Python SciPy stats.kappa4用法及代碼示例
- Python SciPy stats.kstwobign用法及代碼示例
- Python SciPy stats.kstest用法及代碼示例
- Python SciPy stats.kstwo用法及代碼示例
- Python SciPy stats.kstat用法及代碼示例
- Python SciPy stats.ksone用法及代碼示例
- Python SciPy stats.ks_2samp用法及代碼示例
- Python SciPy stats.ks_1samp用法及代碼示例
- Python SciPy stats.anderson用法及代碼示例
- Python SciPy stats.iqr用法及代碼示例
- Python SciPy stats.genpareto用法及代碼示例
- Python SciPy stats.skewnorm用法及代碼示例
- Python SciPy stats.cosine用法及代碼示例
- Python SciPy stats.norminvgauss用法及代碼示例
- Python SciPy stats.directional_stats用法及代碼示例
- Python SciPy stats.invwishart用法及代碼示例
- Python SciPy stats.bartlett用法及代碼示例
- Python SciPy stats.levy_stable用法及代碼示例
- Python SciPy stats.page_trend_test用法及代碼示例
- Python SciPy stats.itemfreq用法及代碼示例
- Python SciPy stats.exponpow用法及代碼示例
注:本文由純淨天空篩選整理自scipy.org大神的英文原創作品 scipy.stats.kendalltau。非經特殊聲明,原始代碼版權歸原作者所有,本譯文未經允許或授權,請勿轉載或複製。
