对数据集进行异常检测及比较简介
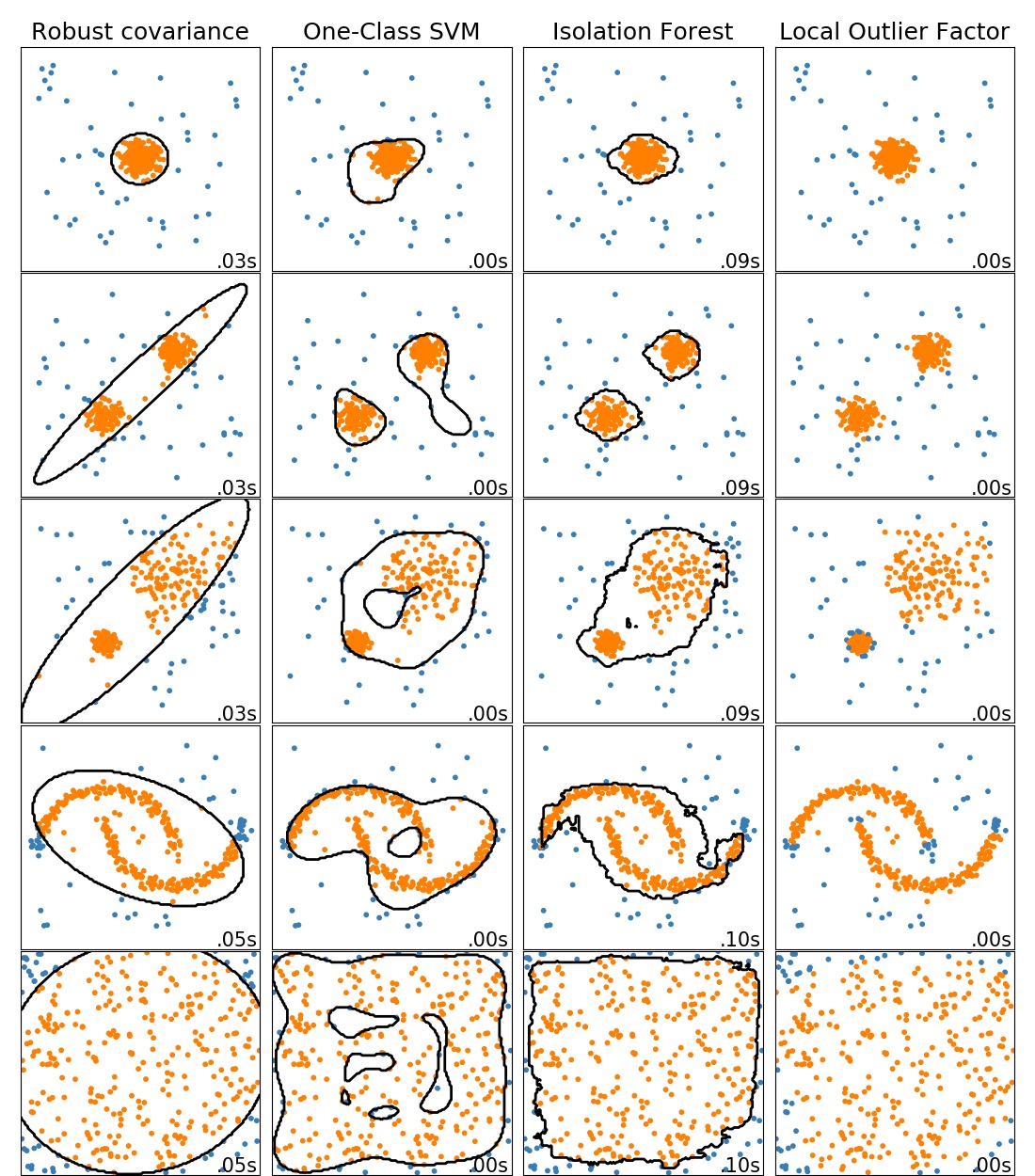
此示例显示了2D数据集上不同异常检测算法的特征。数据集包含一种或两种模式(高密度区域),以说明算法处理多模式数据的能力。
示例中有5个数据集,对于每个数据集,将生成15%的样本作为随机均匀噪声。该比例是为OneClassSVM的nu参数和其他异常值检测算法的污染参数提供的值。离群值和离群值之间的决策边界以黑色显示,但局部离群值因子(LOF)除外,因为当用于离群值检测时,它没有适用于新数据的预测方法。
sklearn.svm.OneClassSVM已知对异常值敏感,因此对于异常值检测效果不佳。当训练集不受异常值污染时,此估计器最适合新颖性检测;而且,在高维空间中检测异常值,或者不对基础数据的分布进行任何假设都是非常具有挑战性的,而One-class SVM在这些情况下可能会得到有用的结果,如果超参数设置合适的话。
sklearn.covariance.EllipticEnvelope假定数据为高斯分布并学习一个椭圆。因此,当数据不是高斯分布的单峰时,性能会降低。但是请注意,这个模型估计对异常值具有鲁棒性。
sklearn.ensemble.IsolationForest和sklearn.neighbors.LocalOutlierFactor对于多模(multi-modal)数据集,效果似乎还不错。sklearn.neighbors.LocalOutlierFactor相对其他模型的优势在第3个数据集有展示,其中两种模式的密度不同。 LOF的局部特性解释了此优势,也就是它仅将一个样本的异常评分与其邻居的评分进行比较。
对于第5个数据集,很难说一个样本比另一个样本异常得多,因为它们均匀分布在超立方体中。除了sklearn.svm.OneClassSVM有点不合适,其他所有估算器都针对这种情况给出了不错的解决方案。在这种情况下,明智的做法是仔细观察样本的异常分数,因为好的模型估计器应该为所有样本分配相似的分数。
另外,此处已手动选择了模型的参数,但实际上需要对其进行进一步调整优化。在没有标记数据的情况下,是非监督学习问题,因此针对不同的实际情况选择模型可能是一个很大的挑战。
注意:尽管这些示例给出了有关算法的一些直觉,但这种直觉可能不适用于非常高维的数据。
代码实现[Python]
# -*- coding: utf-8 -*-
# Author: Alexandre Gramfort
# Albert Thomas
# License: BSD 3 clause
import time
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from sklearn import svm
from sklearn.datasets import make_moons, make_blobs
from sklearn.covariance import EllipticEnvelope
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
print(__doc__)
matplotlib.rcParams['contour.negative_linestyle'] = 'solid'
# Example settings
n_samples = 300
outliers_fraction = 0.15
n_outliers = int(outliers_fraction * n_samples)
n_inliers = n_samples - n_outliers
# define outlier/anomaly detection methods to be compared
anomaly_algorithms = [
("Robust covariance", EllipticEnvelope(contamination=outliers_fraction)),
("One-Class SVM", svm.OneClassSVM(nu=outliers_fraction, kernel="rbf",
gamma=0.1)),
("Isolation Forest", IsolationForest(behaviour='new',
contamination=outliers_fraction,
random_state=42)),
("Local Outlier Factor", LocalOutlierFactor(
n_neighbors=35, contamination=outliers_fraction))]
# 定义数据集,这个给定了5个数据集(Define datasets)
blobs_params = dict(random_state=0, n_samples=n_inliers, n_features=2)
datasets = [
make_blobs(centers=[[0, 0], [0, 0]], cluster_std=0.5,
**blobs_params)[0],
make_blobs(centers=[[2, 2], [-2, -2]], cluster_std=[0.5, 0.5],
**blobs_params)[0],
make_blobs(centers=[[2, 2], [-2, -2]], cluster_std=[1.5, .3],
**blobs_params)[0],
4. * (make_moons(n_samples=n_samples, noise=.05, random_state=0)[0] -
np.array([0.5, 0.25])),
14. * (np.random.RandomState(42).rand(n_samples, 2) - 0.5)]
# 在给定的设置上比较不同分类器模型(Compare given classifiers under given settings)
xx, yy = np.meshgrid(np.linspace(-7, 7, 150),
np.linspace(-7, 7, 150))
plt.figure(figsize=(len(anomaly_algorithms) * 2 + 3, 12.5))
plt.subplots_adjust(left=.02, right=.98, bottom=.001, top=.96, wspace=.05,
hspace=.01)
plot_num = 1
rng = np.random.RandomState(42)
for i_dataset, X in enumerate(datasets):
# Add outliers
X = np.concatenate([X, rng.uniform(low=-6, high=6,
size=(n_outliers, 2))], axis=0)
for name, algorithm in anomaly_algorithms:
t0 = time.time()
algorithm.fit(X)
t1 = time.time()
plt.subplot(len(datasets), len(anomaly_algorithms), plot_num)
if i_dataset == 0:
plt.title(name, size=18)
# fit the data and tag outliers
if name == "Local Outlier Factor":
y_pred = algorithm.fit_predict(X)
else:
y_pred = algorithm.fit(X).predict(X)
# plot the levels lines and the points
if name != "Local Outlier Factor": # LOF does not implement predict
Z = algorithm.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contour(xx, yy, Z, levels=[0], linewidths=2, colors='black')
colors = np.array(['#377eb8', '#ff7f00'])
plt.scatter(X[:, 0], X[:, 1], s=10, color=colors[(y_pred + 1) // 2])
plt.xlim(-7, 7)
plt.ylim(-7, 7)
plt.xticks(())
plt.yticks(())
plt.text(.99, .01, ('%.2fs' % (t1 - t0)).lstrip('0'),
transform=plt.gca().transAxes, size=15,
horizontalalignment='right')
plot_num += 1
plt.show()
代码执行
代码运行时间大约:0分3.491秒。
运行代码输出的图片内容如下:
源码下载
- Python版源码文件: plot_anomaly_comparison.py
- Jupyter Notebook版源码文件: plot_anomaly_comparison.ipynb
