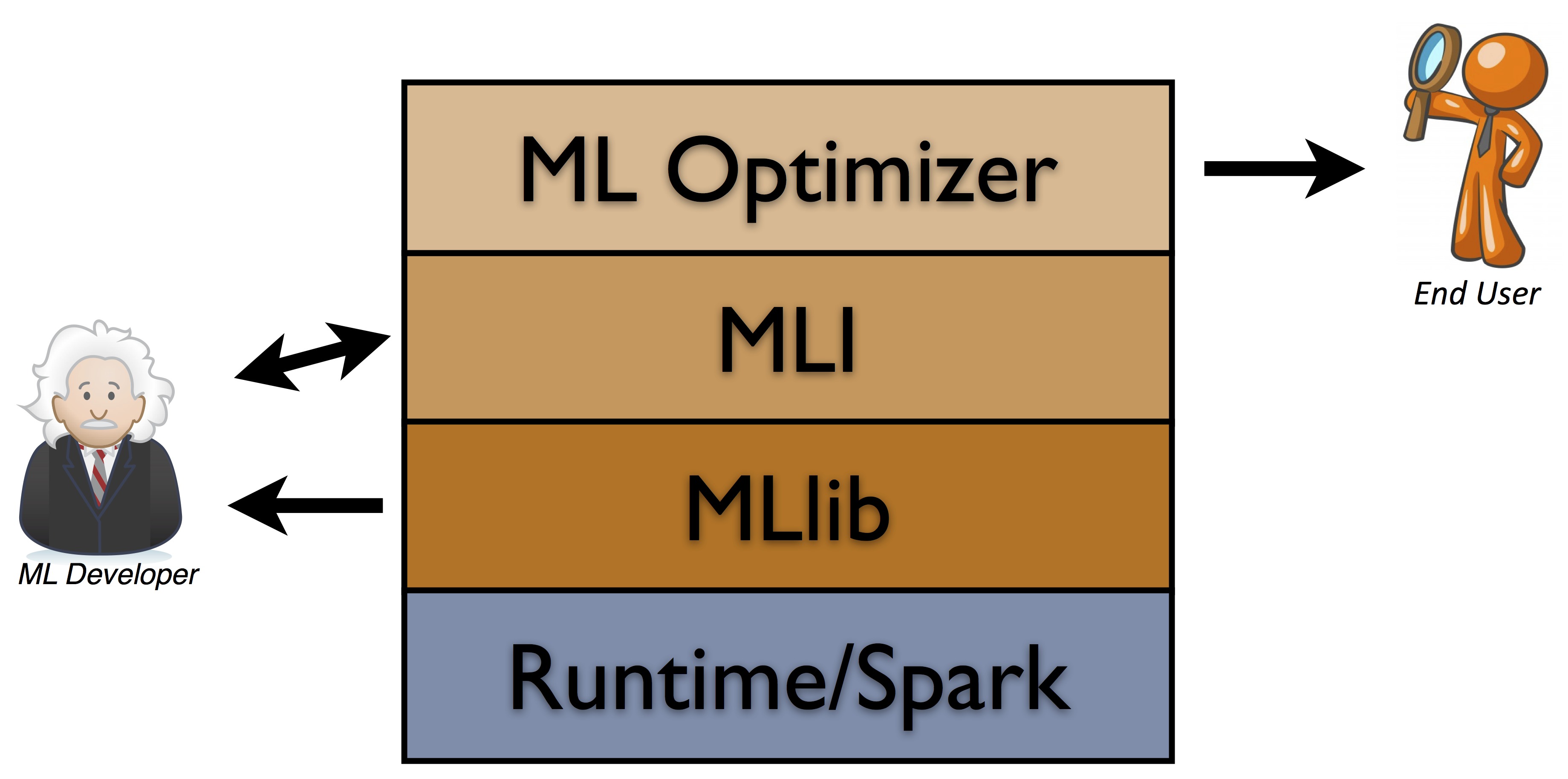
Spark中ml和mllib的主要区别和联系如下:
- ml和mllib都是Spark中的机器学习库,目前常用的机器学习功能2个库都能满足需求。
- spark官方推荐使用ml, 因为ml功能更全面更灵活,未来会主要支持ml,mllib很有可能会被废弃(据说可能是在spark3.0中deprecated)。
- ml主要操作的是DataFrame, 而mllib操作的是RDD,也就是说二者面向的数据集不一样。
- DataFrame和RDD什么关系?DataFrame是Dataset的子集,也就是Dataset[Row], 而DataSet是对RDD的封装,对SQL之类的操作做了很多优化。
- 相比于mllib在RDD提供的基础操作,ml在DataFrame上的抽象级别更高,数据和操作耦合度更低。
- ml中的操作可以使用pipeline, 跟sklearn一样,可以把很多操作(算法/特征提取/特征转换)以管道的形式串起来,然后让数据在这个管道中流动。大家可以脑补一下Linux管道在做任务组合时有多么方便。
- ml中无论是什么模型,都提供了统一的算法操作接口,比如模型训练都是
fit;不像mllib中不同模型会有各种各样的trainXXX。 - mllib在spark2.0之后进入
维护状态, 这个状态通常只修复BUG不增加新功能。
以上就是ml和mllib的主要异同点。下面是ml和mllib逻辑回归的例子,可以对比看一下, 虽然都是模型训练和预测,但是画风很不一样。
mllib中逻辑回归的例子
>>> sparse_data = [
... LabeledPoint(0.0, SparseVector(2, {0: 0.0})),
... LabeledPoint(1.0, SparseVector(2, {1: 1.0})),
... LabeledPoint(0.0, SparseVector(2, {0: 1.0})),
... LabeledPoint(1.0, SparseVector(2, {1: 2.0}))
... ]
>>> lrm = LogisticRegressionWithSGD.train(sc.parallelize(sparse_data), iterations=10)
>>> lrm.predict(array([0.0, 1.0]))
1
>>> lrm.predict(array([1.0, 0.0]))
0
>>> lrm.predict(SparseVector(2, {1: 1.0}))
1
>>> lrm.predict(SparseVector(2, {0: 1.0}))
0
>>> import os, tempfile
>>> path = tempfile.mkdtemp()
>>> lrm.save(sc, path)
>>> sameModel = LogisticRegressionModel.load(sc, path)
>>> sameModel.predict(array([0.0, 1.0]))
1
>>> sameModel.predict(SparseVector(2, {0: 1.0}))
0
>>> from shutil import rmtree
>>> try:
... rmtree(path)
... except:
... pass
>>> multi_class_data = [
... LabeledPoint(0.0, [0.0, 1.0, 0.0]),
... LabeledPoint(1.0, [1.0, 0.0, 0.0]),
... LabeledPoint(2.0, [0.0, 0.0, 1.0])
... ]
>>> data = sc.parallelize(multi_class_data)
>>> mcm = LogisticRegressionWithLBFGS.train(data, iterations=10, numClasses=3)
>>> mcm.predict([0.0, 0.5, 0.0])
0
>>> mcm.predict([0.8, 0.0, 0.0])
1
>>> mcm.predict([0.0, 0.0, 0.3])
2
ml中的逻辑回归的例子
>>> from pyspark.sql import Row
>>> from pyspark.ml.linalg import Vectors
>>> bdf = sc.parallelize([
... Row(label=1.0, weight=2.0, features=Vectors.dense(1.0)),
... Row(label=0.0, weight=2.0, features=Vectors.sparse(1, [], []))]).toDF()
>>> blor = LogisticRegression(maxIter=5, regParam=0.01, weightCol="weight")
>>> blorModel = blor.fit(bdf)
>>> blorModel.coefficients
DenseVector([5.5...])
>>> blorModel.intercept
-2.68...
>>> mdf = sc.parallelize([
... Row(label=1.0, weight=2.0, features=Vectors.dense(1.0)),
... Row(label=0.0, weight=2.0, features=Vectors.sparse(1, [], [])),
... Row(label=2.0, weight=2.0, features=Vectors.dense(3.0))]).toDF()
>>> mlor = LogisticRegression(maxIter=5, regParam=0.01, weightCol="weight",
... family="multinomial")
>>> mlorModel = mlor.fit(mdf)
>>> print(mlorModel.coefficientMatrix)
DenseMatrix([[-2.3...],
[ 0.2...],
[ 2.1... ]])
>>> mlorModel.interceptVector
DenseVector([2.0..., 0.8..., -2.8...])
>>> test0 = sc.parallelize([Row(features=Vectors.dense(-1.0))]).toDF()
>>> result = blorModel.transform(test0).head()
>>> result.prediction
0.0
>>> result.probability
DenseVector([0.99..., 0.00...])
>>> result.rawPrediction
DenseVector([8.22..., -8.22...])
>>> test1 = sc.parallelize([Row(features=Vectors.sparse(1, [0], [1.0]))]).toDF()
>>> blorModel.transform(test1).head().prediction
1.0
>>> blor.setParams("vector")
Traceback (most recent call last):
...
TypeError: Method setParams forces keyword arguments.
>>> lr_path = temp_path + "/lr"
>>> blor.save(lr_path)
>>> lr2 = LogisticRegression.load(lr_path)
>>> lr2.getMaxIter()
5
>>> model_path = temp_path + "/lr_model"
>>> blorModel.save(model_path)
>>> model2 = LogisticRegressionModel.load(model_path)
>>> blorModel.coefficients[0] == model2.coefficients[0]
True
>>> blorModel.intercept == model2.intercept
True
