step_downsample() 創建配方步驟的規範,該步驟將刪除數據集的行,以使特定因子級別中級別的出現次數相等。
用法
step_downsample(
recipe,
...,
under_ratio = 1,
ratio = deprecated(),
role = NA,
trained = FALSE,
column = NULL,
target = NA,
skip = TRUE,
seed = sample.int(10^5, 1),
id = rand_id("downsample")
)參數
- recipe
-
一個菜譜對象。該步驟將添加到此配方的操作序列中。
- ...
-
一個或多個選擇器函數用於選擇使用哪個變量對數據進行采樣。有關更多詳細信息,請參閱
selections()。選擇應產生單因子變量。對於tidy方法,當前未使用這些。 - under_ratio
-
minority-to-majority 頻率比率的數值。默認值 (1) 意味著所有其他級別都會向下采樣,以具有與最少出現的級別相同的頻率。值為 2 意味著多數級別的行數(最多)(大約)是少數級別的行數的兩倍。
- ratio
-
已棄用的參數;與
under_ratio相同 - role
-
由於沒有創建新變量,因此此步驟未使用。
- trained
-
指示預處理數量是否已估計的邏輯。
- column
-
將由
...選擇器(最終)填充的變量名稱的字符串。 - target
-
將用於二次采樣的整數。這不應由用戶設置,並將由
prep填充。 - skip
-
一個合乎邏輯的。當
bake()烘焙食譜時是否應該跳過此步驟?雖然所有操作都是在prep()運行時烘焙的,但某些操作可能無法對新數據進行(例如處理結果變量)。使用skip = TRUE時應小心,因為它可能會影響後續操作的計算。 - seed
-
下采樣時用作種子的整數。
- id
-
該步驟特有的字符串,用於標識它。
細節
下采樣旨在單獨在訓練集上執行。因此,默認值為 skip = TRUE 。
如果用於定義抽樣的因子變量中存在缺失值,則將以與其他因子水平抽樣相同的方式隨機選擇缺失數據。缺失值不用於確定少數級別的數據量
對於因子水平出現頻率與少數水平相同的任何數據,所有數據都將被保留。
數據中的所有列均由 juice() 和 bake() 采樣並返回。
請記住,步驟中下采樣的位置可能會產生影響。例如,如果居中和縮放,則不清楚這些操作應該在刪除行之前還是之後進行。
整理
當您tidy()此步驟時,將返回包含列terms(選擇的選擇器或變量)的tibble。
箱重
此步驟執行可以利用案例權重的無監督操作。要使用它們,請參閱 recipes::case_weights 中的文檔和 tidymodels.org 中的示例。
也可以看看
under-sampling 的其他步驟:step_nearmiss()、step_tomek()
例子
library(recipes)
library(modeldata)
data(hpc_data)
hpc_data0 <- hpc_data %>%
select(-protocol, -day)
orig <- count(hpc_data0, class, name = "orig")
orig
#> # A tibble: 4 × 2
#> class orig
#> <fct> <int>
#> 1 VF 2211
#> 2 F 1347
#> 3 M 514
#> 4 L 259
up_rec <- recipe(class ~ ., data = hpc_data0) %>%
# Bring the majority levels down to about 1000 each
# 1000/259 is approx 3.862
step_downsample(class, under_ratio = 3.862) %>%
prep()
training <- up_rec %>%
bake(new_data = NULL) %>%
count(class, name = "training")
training
#> # A tibble: 4 × 2
#> class training
#> <fct> <int>
#> 1 VF 1000
#> 2 F 1000
#> 3 M 514
#> 4 L 259
# Since `skip` defaults to TRUE, baking the step has no effect
baked <- up_rec %>%
bake(new_data = hpc_data0) %>%
count(class, name = "baked")
baked
#> # A tibble: 4 × 2
#> class baked
#> <fct> <int>
#> 1 VF 2211
#> 2 F 1347
#> 3 M 514
#> 4 L 259
# Note that if the original data contained more rows than the
# target n (= ratio * majority_n), the data are left alone:
orig %>%
left_join(training, by = "class") %>%
left_join(baked, by = "class")
#> # A tibble: 4 × 4
#> class orig training baked
#> <fct> <int> <int> <int>
#> 1 VF 2211 1000 2211
#> 2 F 1347 1000 1347
#> 3 M 514 514 514
#> 4 L 259 259 259
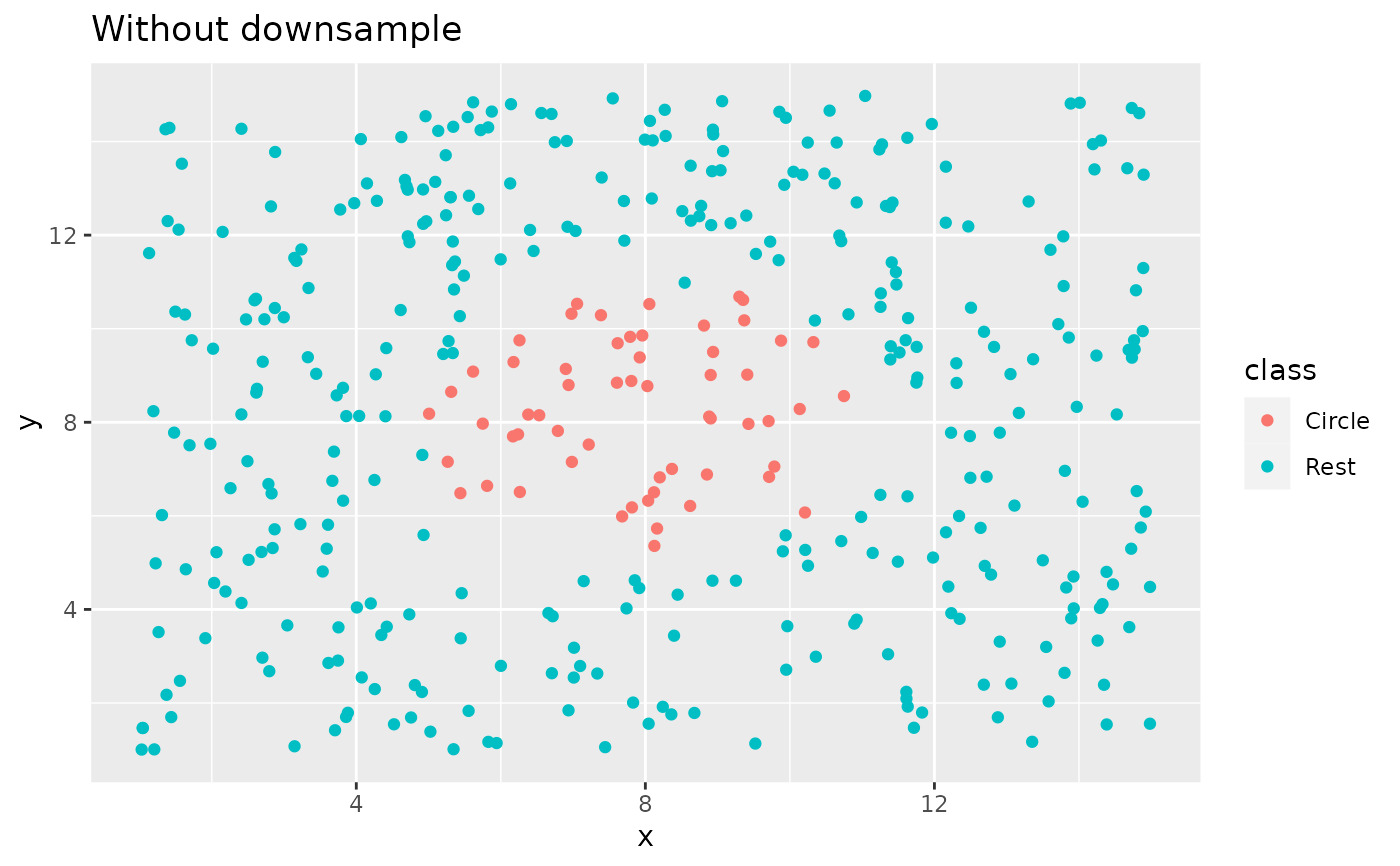
library(ggplot2)
ggplot(circle_example, aes(x, y, color = class)) +
geom_point() +
labs(title = "Without downsample")
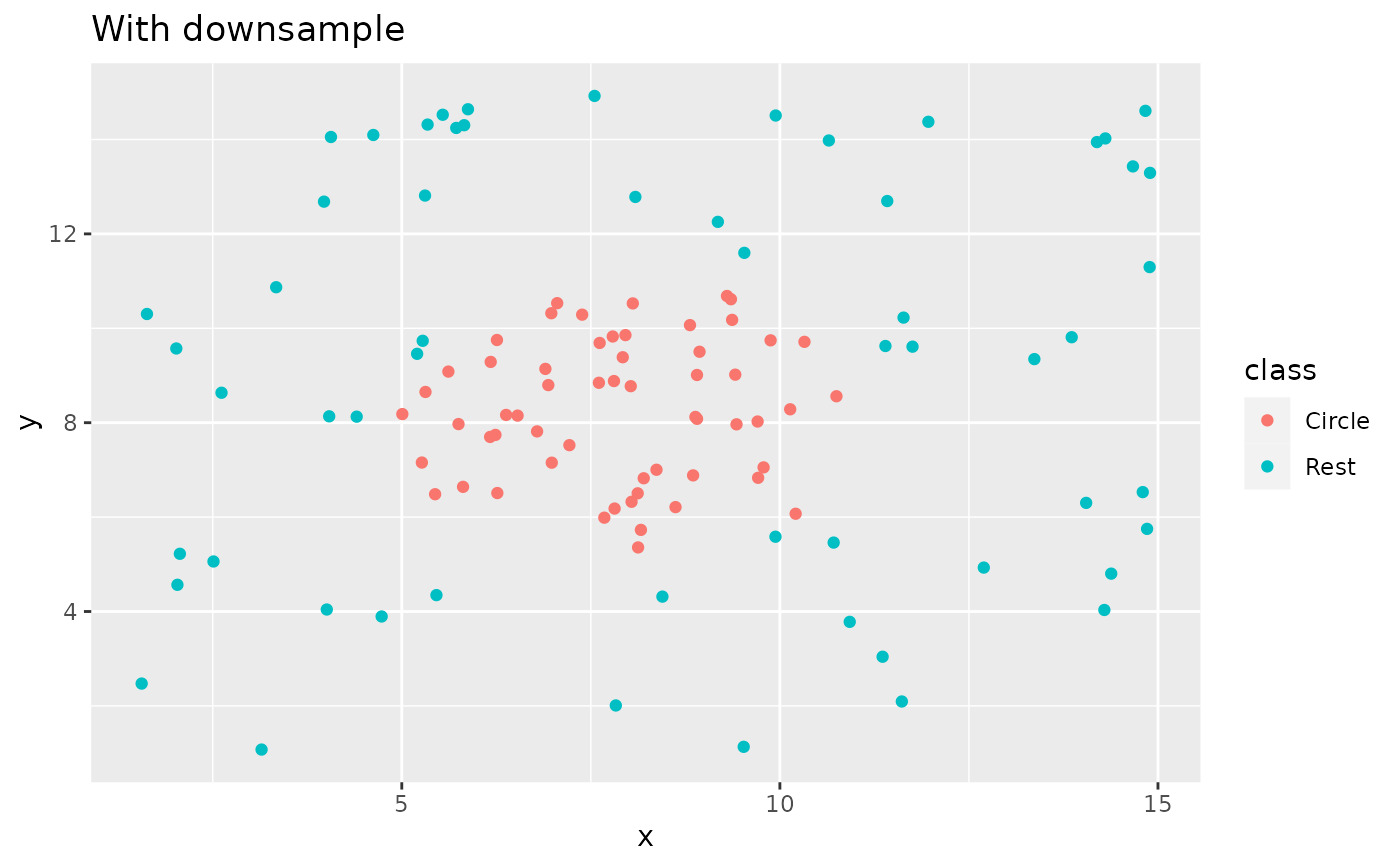
 recipe(class ~ x + y, data = circle_example) %>%
step_downsample(class) %>%
prep() %>%
bake(new_data = NULL) %>%
ggplot(aes(x, y, color = class)) +
geom_point() +
labs(title = "With downsample")
recipe(class ~ x + y, data = circle_example) %>%
step_downsample(class) %>%
prep() %>%
bake(new_data = NULL) %>%
ggplot(aes(x, y, color = class)) +
geom_point() +
labs(title = "With downsample")
相關用法
- R themis step_smote 應用SMOTE算法
- R themis step_smotenc 應用 SMOTENC 算法
- R themis step_tomek 刪除 Tomek 的鏈接
- R themis step_rose 應用ROSE算法
- R themis step_upsample 基於因子變量對數據集進行上采樣
- R themis step_bsmote 應用邊界-SMOTE 算法
- R themis step_nearmiss 刪除其他類附近的點
- R themis step_adasyn 應用自適應合成算法
- R themis smotenc SMOTENC算法
- R themis smote SMOTE算法
- R themis tomek 刪除 Tomek 的鏈接
- R themis bsmote 邊界-SMOTE算法
- R themis nearmiss 刪除其他類附近的點
- R themis adasyn 自適應合成算法
- R update_PACKAGES 更新現有的 PACKAGES 文件
- R textrecipes tokenlist 創建令牌對象
- R print.via.format 打印實用程序
- R tibble tibble 構建 DataFrame 架
- R tidyr separate_rows 將折疊的列分成多行
- R textrecipes step_lemma 標記變量的詞形還原
- R textrecipes show_tokens 顯示配方的令牌輸出
- R tidyr extract 使用正則表達式組將字符列提取為多列
- R prepare_Rd 準備用於渲染的解析 Rd 對象
- R tidyr chop 砍伐和砍伐
- R tidyr pivot_longer_spec 使用規範將數據從寬轉為長
注:本文由純淨天空篩選整理自等大神的英文原創作品 Down-Sample a Data Set Based on a Factor Variable。非經特殊聲明,原始代碼版權歸原作者所有,本譯文未經允許或授權,請勿轉載或複製。
