scream() 确保 data 的结构与原型 ptype 相同。在底层,使用 vctrs::vec_cast() ,它将 data 的每一列转换为与 ptype 中相应列相同的类型。
该铸造强制执行许多重要的结构检查,包括但不限于:
-
数据类 - 检查
data中每列的类是否与ptype中相应列的类相同。 -
新级别 - 检查
data中的因子列与ptype列相比是否没有任何新级别。如果有新级别,则会发出警告,并将它们强制为NA。此检查是可选的,可以使用allow_novel_levels = TRUE关闭。 -
级别恢复 - 检查
data中的因子列与ptype列相比是否缺少任何因子级别。如果存在缺失的关卡,则会将其恢复。
参数
- data
-
包含要检查其结构的新数据的 DataFrame 。
- ptype
-
将
data转换为的 DataFrame 原型。这通常是训练集的 0 行切片。 - allow_novel_levels
-
是否应该允许
data中的新因子水平?最安全的方法是默认方法,当发现新级别时会发出警告,并将它们强制为NA值。将此参数设置为TRUE将忽略所有新级别。这个论点不适用于有序因子。有序因子中不允许出现新颖的级别,因为级别排序是该类型的关键部分。
细节
scream() 由forge() 在shrink() 之后但在实际处理完成之前调用。一般来说,你不需要直接调用scream(),因为forge()会为你做这件事。
如果 scream() 用作独立函数,则最好在其之前调用 shrink(),因为 scream() 中没有检查来确保 data 中实际存在所有必需的列名称。这些检查存在于 shrink() 中。
因子水平
scream() 尝试通过恢复缺失的因子水平并警告新水平来提供帮助。下图概述了从 data 中的列强制到 ptype 中的列时 scream() 如何处理因子级别。
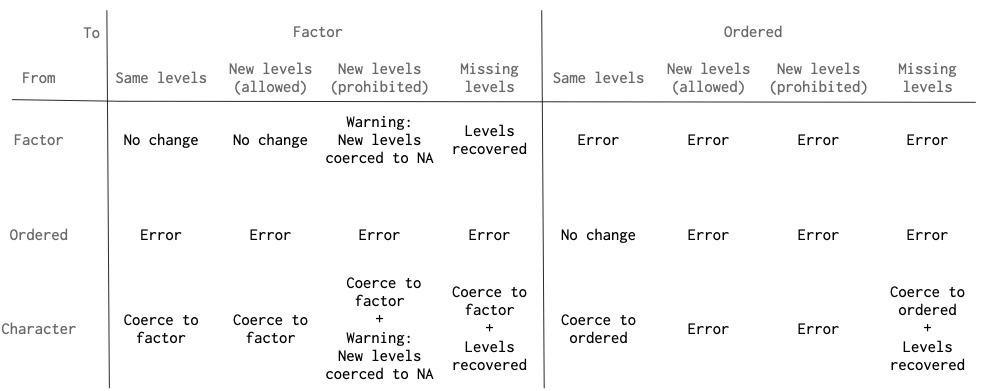
请注意,有序因子处理比因子处理严格得多。 data 中的有序因子必须与 ptype 中的有序因子具有完全相同的级别。
例子
# ---------------------------------------------------------------------------
# Setup
train <- iris[1:100, ]
test <- iris[101:150, ]
# mold() is run at model fit time
# and a formula preprocessing blueprint is recorded
x <- mold(log(Sepal.Width) ~ Species, train)
# Inside the result of mold() are the prototype tibbles
# for the predictors and the outcomes
ptype_pred <- x$blueprint$ptypes$predictors
ptype_out <- x$blueprint$ptypes$outcomes
# ---------------------------------------------------------------------------
# shrink() / scream()
# Pass the test data, along with a prototype, to
# shrink() to extract the prototype columns
test_shrunk <- shrink(test, ptype_pred)
# Now pass that to scream() to perform validation checks
# If no warnings / errors are thrown, the checks were
# successful!
scream(test_shrunk, ptype_pred)
#> # A tibble: 50 × 1
#> Species
#> <fct>
#> 1 virginica
#> 2 virginica
#> 3 virginica
#> 4 virginica
#> 5 virginica
#> 6 virginica
#> 7 virginica
#> 8 virginica
#> 9 virginica
#> 10 virginica
#> # ℹ 40 more rows
# ---------------------------------------------------------------------------
# Outcomes
# To also extract the outcomes, use the outcome prototype
test_outcome <- shrink(test, ptype_out)
scream(test_outcome, ptype_out)
#> # A tibble: 50 × 1
#> Sepal.Width
#> <dbl>
#> 1 3.3
#> 2 2.7
#> 3 3
#> 4 2.9
#> 5 3
#> 6 3
#> 7 2.5
#> 8 2.9
#> 9 2.5
#> 10 3.6
#> # ℹ 40 more rows
# ---------------------------------------------------------------------------
# Casting
# scream() uses vctrs::vec_cast() to intelligently convert
# new data to the prototype automatically. This means
# it can automatically perform certain conversions, like
# coercing character columns to factors.
test2 <- test
test2$Species <- as.character(test2$Species)
test2_shrunk <- shrink(test2, ptype_pred)
scream(test2_shrunk, ptype_pred)
#> # A tibble: 50 × 1
#> Species
#> <fct>
#> 1 virginica
#> 2 virginica
#> 3 virginica
#> 4 virginica
#> 5 virginica
#> 6 virginica
#> 7 virginica
#> 8 virginica
#> 9 virginica
#> 10 virginica
#> # ℹ 40 more rows
# It can also recover missing factor levels.
# For example, it is plausible that the test data only had the
# "virginica" level
test3 <- test
test3$Species <- factor(test3$Species, levels = "virginica")
test3_shrunk <- shrink(test3, ptype_pred)
test3_fixed <- scream(test3_shrunk, ptype_pred)
# scream() recovered the missing levels
levels(test3_fixed$Species)
#> [1] "setosa" "versicolor" "virginica"
# ---------------------------------------------------------------------------
# Novel levels
# When novel levels with any data are present in `data`, the default
# is to coerce them to `NA` values with a warning.
test4 <- test
test4$Species <- as.character(test4$Species)
test4$Species[1] <- "new_level"
test4$Species <- factor(
test4$Species,
levels = c(levels(test$Species), "new_level")
)
test4 <- shrink(test4, ptype_pred)
# Warning is thrown
test4_removed <- scream(test4, ptype_pred)
#> Warning: Novel levels found in column 'Species': 'new_level'. The levels have been removed, and values have been coerced to 'NA'.
# Novel level is removed
levels(test4_removed$Species)
#> [1] "setosa" "versicolor" "virginica"
# No warning is thrown
test4_kept <- scream(test4, ptype_pred, allow_novel_levels = TRUE)
# Novel level is kept
levels(test4_kept$Species)
#> [1] "setosa" "versicolor" "virginica" "new_level"
相关用法
- R hardhat standardize 标准化结果
- R hardhat shrink 仅对所需列进行子集化
- R hardhat spruce-multiple 完善多结果预测
- R hardhat validate_prediction_size 确保预测具有正确的行数
- R hardhat default_recipe_blueprint 默认配方蓝图
- R hardhat is_blueprint x 是预处理蓝图吗?
- R hardhat validate_column_names 确保数据包含所需的列名
- R hardhat default_formula_blueprint 默认公式蓝图
- R hardhat update_blueprint 更新预处理蓝图
- R hardhat weighted_table 加权表
- R hardhat validate_outcomes_are_univariate 确保结果是单变量
- R hardhat get_levels 从 DataFrame 中提取因子水平
- R hardhat add_intercept_column 向数据添加截距列
- R hardhat is_frequency_weights x 是频率权重向量吗?
- R hardhat model_offset 提取模型偏移
- R hardhat model_matrix 构建设计矩阵
- R hardhat is_importance_weights x 是重要性权重向量吗?
- R hardhat run-mold 根据蓝图 Mold()
- R hardhat get_data_classes 从 DataFrame 或矩阵中提取数据类
- R hardhat fct_encode_one_hot 将一个因子编码为 one-hot 指标矩阵
- R hardhat new_frequency_weights 构建频率权重向量
- R hardhat validate_no_formula_duplication 确保公式中不出现重复项
- R hardhat default_xy_blueprint 默认 XY 蓝图
- R hardhat validate_outcomes_are_numeric 确保结果都是数字
- R hardhat frequency_weights 频率权重
注:本文由纯净天空筛选整理自Davis Vaughan等大神的英文原创作品 ? Scream.。非经特殊声明,原始代码版权归原作者所有,本译文未经允许或授权,请勿转载或复制。
