bart() 定义了一个树集成模型,该模型使用贝叶斯分析来组装集成。该函数可以拟合分类和回归模型。
拟合该模型的方法有多种,通过设置模型引擎来选择估计方法。下面列出了该模型的引擎特定页面。
1 默认引擎。有关如何操作的更多信息防风草用于建模的是https://www.tidymodels.org/.
用法
bart(
mode = "unknown",
engine = "dbarts",
trees = NULL,
prior_terminal_node_coef = NULL,
prior_terminal_node_expo = NULL,
prior_outcome_range = NULL
)参数
- mode
-
预测结果模式的单个字符串。此模型的可能值为"unknown"、"regression" 或"classification"。
- engine
-
指定用于拟合的计算引擎的单个字符串。
- trees
-
集合中包含的树数的整数。
- prior_terminal_node_coef
-
节点是终端节点的先验概率的系数。值通常介于 0 和 1 之间,默认值为 0.95。这会影响基线概率;数字越小,总体概率就越大。请参阅下面的详细信息。
- prior_terminal_node_expo
-
节点是终端节点的先验概率的指数。值通常为非负数,默认值为 2。这会影响先验概率随着树深度的增加而降低的速率。值越大,树越深的可能性就越小。
- prior_outcome_range
-
一个正值,定义预测结果在一定范围内的先验宽度。对于回归来说,它与观察到的数据范围有关;先验是由数据观测范围定义的高斯分布的标准差数。对于分类,它被定义为+/-3的范围(假设在logit标度上)。默认值为 2。
细节
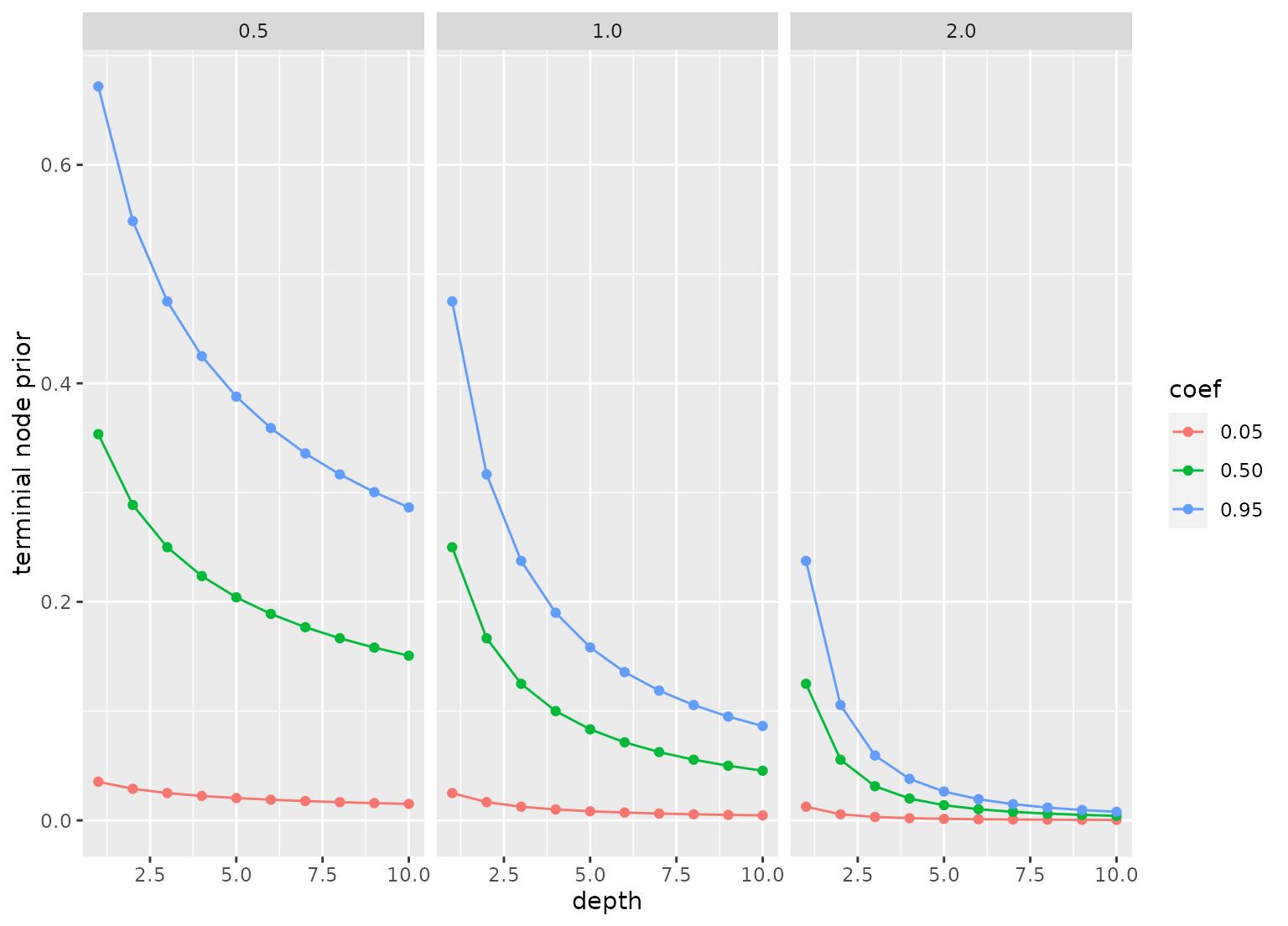
终端节点概率的先验表示为prior = a * (1 + d)^(-b),其中d是节点的深度,a是prior_terminal_node_coef,b是prior_terminal_node_expo。请参阅下面的示例部分,了解这些参数的不同值的终端节点的先验概率的示例图。
此函数仅定义正在拟合的模型类型。一旦指定了引擎,也就定义了拟合模型的方法。有关设置引擎的更多信息,包括如何设置引擎参数,请参阅set_engine()。
在 fit() 函数与数据一起使用之前,模型不会经过训练或拟合。
此函数中除 mode 和 engine 之外的每个参数都被捕获为 quosures 。要以编程方式传递值,请使用injection operator,如下所示:
value <- 1
bart(argument = !!value)例子
show_engines("bart")
#> # A tibble: 2 × 2
#> engine mode
#> <chr> <chr>
#> 1 dbarts classification
#> 2 dbarts regression
bart(mode = "regression", trees = 5)
#> BART Model Specification (regression)
#>
#> Main Arguments:
#> trees = 5
#>
#> Computational engine: dbarts
#>
# ------------------------------------------------------------------------------
# Examples for terminal node prior
library(ggplot2)
library(dplyr)
#>
#> Attaching package: ‘dplyr’
#> The following objects are masked from ‘package:stats’:
#>
#> filter, lag
#> The following objects are masked from ‘package:base’:
#>
#> intersect, setdiff, setequal, union
prior_test <- function(coef = 0.95, expo = 2, depths = 1:10) {
tidyr::crossing(coef = coef, expo = expo, depth = depths) %>%
mutate(
`terminial node prior` = coef * (1 + depth)^(-expo),
coef = format(coef),
expo = format(expo))
}
prior_test(coef = c(0.05, 0.5, .95), expo = c(1/2, 1, 2)) %>%
ggplot(aes(depth, `terminial node prior`, col = coef)) +
geom_line() +
geom_point() +
facet_wrap(~ expo)
相关用法
- R parsnip boost_tree 增强树
- R parsnip logistic_reg 逻辑回归
- R parsnip predict.model_fit 模型预测
- R parsnip linear_reg 线性回归
- R parsnip C5_rules C5.0 基于规则的分类模型
- R parsnip set_engine 声明计算引擎和特定参数
- R parsnip condense_control 将控制对象压缩为更小的控制对象
- R parsnip control_parsnip 控制拟合函数
- R parsnip augment 通过预测增强数据
- R parsnip repair_call 修复模型调用对象
- R parsnip dot-model_param_name_key 翻译模型调整参数的名称
- R parsnip glm_grouped 将数据集中的分组二项式结果与个案权重拟合
- R parsnip rule_fit 规则拟合模型
- R parsnip svm_rbf 径向基函数支持向量机
- R parsnip set_args 更改模型规范的元素
- R parsnip translate 解决计算引擎的模型规范
- R parsnip max_mtry_formula 根据公式确定 mtry 的最大值。此函数可能会根据公式和数据集限制 mtry 的值。对于生存和/或多变量模型来说,这是一种安全的方法。
- R parsnip svm_linear 线性支持向量机
- R parsnip set_new_model 注册模型的工具
- R parsnip rand_forest 随机森林
- R parsnip mlp 单层神经网络
- R parsnip nearest_neighbor K-最近邻
- R parsnip parsnip_update 更新型号规格
- R parsnip fit 将模型规范拟合到数据集
- R parsnip add_rowindex 将一列行号添加到 DataFrame
注:本文由纯净天空筛选整理自Max Kuhn等大神的英文原创作品 Bayesian additive regression trees (BART)。非经特殊声明,原始代码版权归原作者所有,本译文未经允许或授权,请勿转载或复制。
