bart() 定義了一個樹集成模型,該模型使用貝葉斯分析來組裝集成。該函數可以擬合分類和回歸模型。
擬合該模型的方法有多種,通過設置模型引擎來選擇估計方法。下麵列出了該模型的引擎特定頁麵。
1 默認引擎。有關如何操作的更多信息防風草用於建模的是https://www.tidymodels.org/.
用法
bart(
mode = "unknown",
engine = "dbarts",
trees = NULL,
prior_terminal_node_coef = NULL,
prior_terminal_node_expo = NULL,
prior_outcome_range = NULL
)參數
- mode
-
預測結果模式的單個字符串。此模型的可能值為"unknown"、"regression" 或"classification"。
- engine
-
指定用於擬合的計算引擎的單個字符串。
- trees
-
集合中包含的樹數的整數。
- prior_terminal_node_coef
-
節點是終端節點的先驗概率的係數。值通常介於 0 和 1 之間,默認值為 0.95。這會影響基線概率;數字越小,總體概率就越大。請參閱下麵的詳細信息。
- prior_terminal_node_expo
-
節點是終端節點的先驗概率的指數。值通常為非負數,默認值為 2。這會影響先驗概率隨著樹深度的增加而降低的速率。值越大,樹越深的可能性就越小。
- prior_outcome_range
-
一個正值,定義預測結果在一定範圍內的先驗寬度。對於回歸來說,它與觀察到的數據範圍有關;先驗是由數據觀測範圍定義的高斯分布的標準差數。對於分類,它被定義為+/-3的範圍(假設在logit標度上)。默認值為 2。
細節
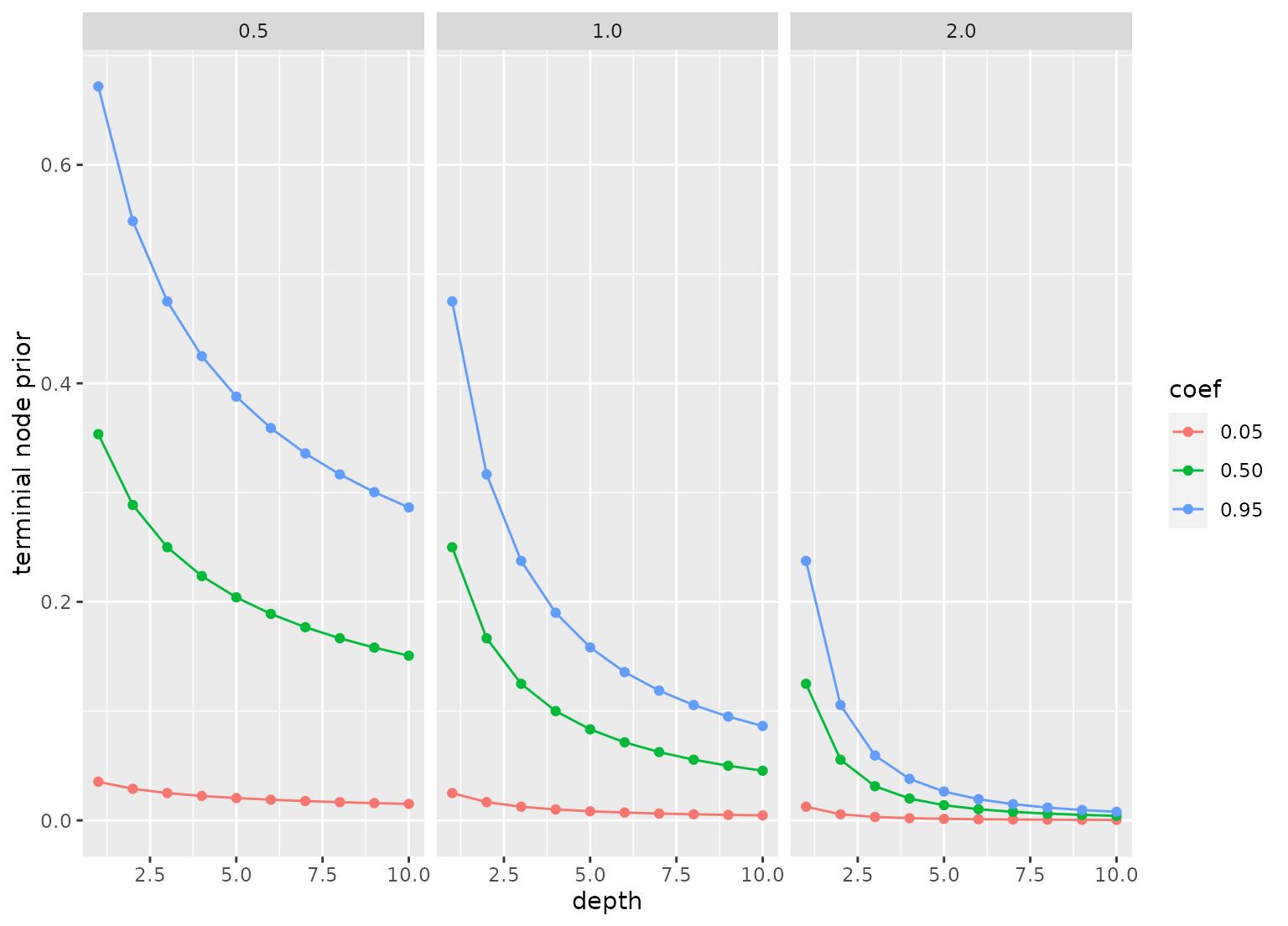
終端節點概率的先驗表示為prior = a * (1 + d)^(-b),其中d是節點的深度,a是prior_terminal_node_coef,b是prior_terminal_node_expo。請參閱下麵的示例部分,了解這些參數的不同值的終端節點的先驗概率的示例圖。
此函數僅定義正在擬合的模型類型。一旦指定了引擎,也就定義了擬合模型的方法。有關設置引擎的更多信息,包括如何設置引擎參數,請參閱set_engine()。
在 fit() 函數與數據一起使用之前,模型不會經過訓練或擬合。
此函數中除 mode 和 engine 之外的每個參數都被捕獲為 quosures 。要以編程方式傳遞值,請使用injection operator,如下所示:
value <- 1
bart(argument = !!value)例子
show_engines("bart")
#> # A tibble: 2 × 2
#> engine mode
#> <chr> <chr>
#> 1 dbarts classification
#> 2 dbarts regression
bart(mode = "regression", trees = 5)
#> BART Model Specification (regression)
#>
#> Main Arguments:
#> trees = 5
#>
#> Computational engine: dbarts
#>
# ------------------------------------------------------------------------------
# Examples for terminal node prior
library(ggplot2)
library(dplyr)
#>
#> Attaching package: ‘dplyr’
#> The following objects are masked from ‘package:stats’:
#>
#> filter, lag
#> The following objects are masked from ‘package:base’:
#>
#> intersect, setdiff, setequal, union
prior_test <- function(coef = 0.95, expo = 2, depths = 1:10) {
tidyr::crossing(coef = coef, expo = expo, depth = depths) %>%
mutate(
`terminial node prior` = coef * (1 + depth)^(-expo),
coef = format(coef),
expo = format(expo))
}
prior_test(coef = c(0.05, 0.5, .95), expo = c(1/2, 1, 2)) %>%
ggplot(aes(depth, `terminial node prior`, col = coef)) +
geom_line() +
geom_point() +
facet_wrap(~ expo)
相關用法
- R parsnip boost_tree 增強樹
- R parsnip logistic_reg 邏輯回歸
- R parsnip predict.model_fit 模型預測
- R parsnip linear_reg 線性回歸
- R parsnip C5_rules C5.0 基於規則的分類模型
- R parsnip set_engine 聲明計算引擎和特定參數
- R parsnip condense_control 將控製對象壓縮為更小的控製對象
- R parsnip control_parsnip 控製擬合函數
- R parsnip augment 通過預測增強數據
- R parsnip repair_call 修複模型調用對象
- R parsnip dot-model_param_name_key 翻譯模型調整參數的名稱
- R parsnip glm_grouped 將數據集中的分組二項式結果與個案權重擬合
- R parsnip rule_fit 規則擬合模型
- R parsnip svm_rbf 徑向基函數支持向量機
- R parsnip set_args 更改模型規範的元素
- R parsnip translate 解決計算引擎的模型規範
- R parsnip max_mtry_formula 根據公式確定 mtry 的最大值。此函數可能會根據公式和數據集限製 mtry 的值。對於生存和/或多變量模型來說,這是一種安全的方法。
- R parsnip svm_linear 線性支持向量機
- R parsnip set_new_model 注冊模型的工具
- R parsnip rand_forest 隨機森林
- R parsnip mlp 單層神經網絡
- R parsnip nearest_neighbor K-最近鄰
- R parsnip parsnip_update 更新型號規格
- R parsnip fit 將模型規範擬合到數據集
- R parsnip add_rowindex 將一列行號添加到 DataFrame
注:本文由純淨天空篩選整理自Max Kuhn等大神的英文原創作品 Bayesian additive regression trees (BART)。非經特殊聲明,原始代碼版權歸原作者所有,本譯文未經允許或授權,請勿轉載或複製。
