step_umap() 創建配方步驟的規範,將一組函數投射到更小的空間中。
用法
step_umap(
recipe,
...,
role = "predictor",
trained = FALSE,
outcome = NULL,
neighbors = 15,
num_comp = 2,
min_dist = 0.01,
metric = "euclidean",
learn_rate = 1,
epochs = NULL,
options = list(verbose = FALSE, n_threads = 1),
seed = sample(10^5, 2),
prefix = "UMAP",
keep_original_cols = FALSE,
retain = deprecated(),
object = NULL,
skip = FALSE,
id = rand_id("umap")
)參數
- recipe
-
一個菜譜對象。該步驟將添加到此配方的操作序列中。
- ...
-
一個或多個選擇器函數用於為此步驟選擇變量。有關更多詳細信息,請參閱
selections()。 - role
-
對於此步驟創建的模型項,應為其分配什麽分析角色?默認情況下,此步驟根據原始變量創建的新列將用作模型中的預測變量。
- trained
-
指示預處理數量是否已估計的邏輯。
- outcome
-
調用
vars以指定哪個變量用作編碼過程的結果(如果有)。 - neighbors
-
用於構造目標單純形集的最近鄰數的整數。如果
neighbors大於數據點數,則使用較小的值。 - num_comp
-
UMAP 組件數量的整數。如果
num_comp大於所選列的數量減一,則使用較小的值。 - min_dist
-
嵌入點之間的有效最小距離。
- metric
-
字符,用於查找最近鄰居的距離度量類型。有關更多詳細信息,請參閱
uwot::umap()。默認為"euclidean"。 - learn_rate
-
優化過程的學習率的正數。
- epochs
-
鄰居優化的迭代次數。有關更多詳細信息,請參閱
uwot::umap()。 - options
-
要傳遞給
uwot::umap()的選項列表。不應在此處傳遞參數X、n_neighbors、n_components、min_dist、n_epochs、ret_model和learning_rate。默認情況下,設置verbose和n_threads。 - seed
-
用於控製數值方法使用的隨機數的兩個整數。默認值從主會話的數字流中提取,如果在調用
prep()或bake()之前設置種子,則將給出可重現的結果。 - prefix
-
生成的新變量的前綴字符串。請參閱下麵的注釋。
- keep_original_cols
-
將原始變量保留在輸出中的邏輯。默認為
FALSE。 - retain
-
使用
keep_original_cols來指定是否應與新的嵌入變量一起保留原始預測變量。 - object
-
定義編碼的對象。在
recipes::prep()訓練該步驟之前,這是NULL。 - skip
-
一個合乎邏輯的。當
bake()烘焙食譜時是否應該跳過此步驟?雖然所有操作都是在prep()運行時烘焙的,但某些操作可能無法對新數據進行(例如處理結果變量)。使用skip = TRUE時應小心,因為它可能會影響後續操作的計算。 - id
-
該步驟特有的字符串,用於標識它。
細節
UMAP 是統一流形逼近和投影的縮寫,是一種非線性降維技術,可找到數據的局部低維表示。它可以在無人監督的情況下運行,也可以使用不同類型的結果數據(例如數字、因子等)進行監督。
參數 num_comp 控製將保留的組件數量(用於派生組件的原始變量將從數據中刪除)。新組件的名稱以 prefix 和一係列數字開頭。變量名稱用零填充。例如,如果 num_comp < 10 ,它們的名稱將為 UMAP1 - UMAP9 。如果是 num_comp = 101 ,則名稱將為 UMAP1 - UMAP101 。
整理
當您tidy()此步驟時,將返回包含列terms(選擇的選擇器或變量)的tibble。
調整參數
此步驟有 5 個調整參數:
-
num_comp: # 組件(類型:整數,默認值:2) -
neighbors: # 最近鄰居(類型:整數,默認值:15) -
min_dist:點之間的最小距離(類型:double,默認值:0.01) -
learn_rate:學習率(類型:double,默認值:1) -
epochs: # Epochs(類型:整數,默認值:NULL)
保存準備好的配方對象
當保存以供另一個 R 會話使用時,此配方步驟可能需要本機序列化。要了解有關準備菜譜序列化的更多信息,請參閱bundle 包。
參考
麥金尼斯,L.,&希利,J.(2018)。 UMAP:統一流形逼近和降維投影。 https://arxiv.org/abs/1802.03426。
“UMAP 的工作原理”https://umap-learn.readthedocs.io/en/latest/how_umap_works.html
例子
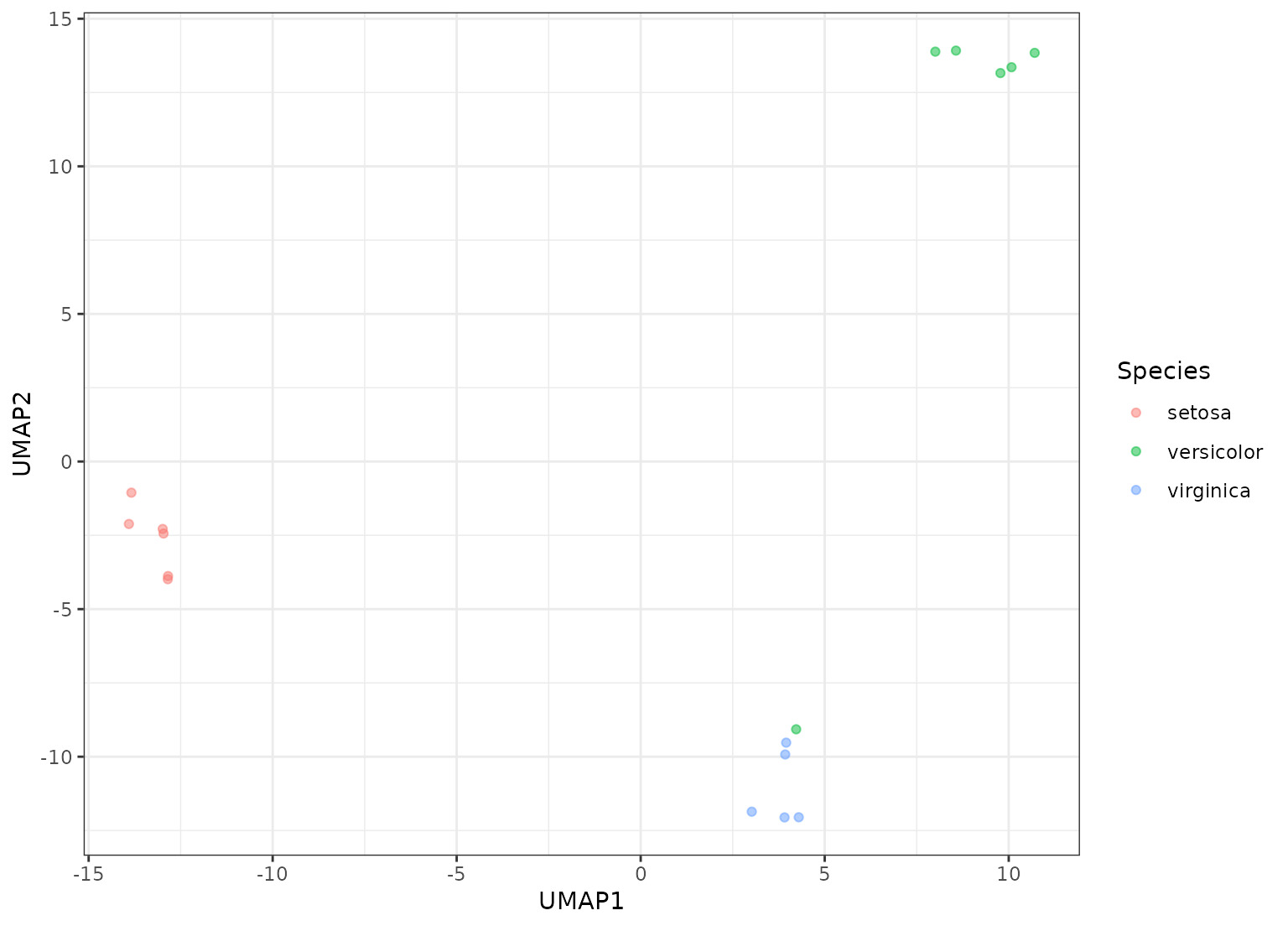
library(recipes)
library(ggplot2)
split <- seq.int(1, 150, by = 9)
tr <- iris[-split, ]
te <- iris[split, ]
set.seed(11)
supervised <-
recipe(Species ~ ., data = tr) %>%
step_center(all_predictors()) %>%
step_scale(all_predictors()) %>%
step_umap(all_predictors(), outcome = vars(Species), num_comp = 2) %>%
prep(training = tr)
theme_set(theme_bw())
bake(supervised, new_data = te, Species, starts_with("umap")) %>%
ggplot(aes(x = UMAP1, y = UMAP2, col = Species)) +
geom_point(alpha = .5)
相關用法
- R embed step_pca_truncated 截斷的 PCA 信號提取
- R embed step_lencode_glm 使用似然編碼將監督因子轉換為線性函數
- R embed step_pca_sparse 稀疏PCA信號提取
- R embed step_lencode_bayes 使用貝葉斯似然編碼將監督因子轉換為線性函數
- R embed step_collapse_stringdist 使用 stringdist 的折疊因子級別
- R embed step_collapse_cart 因子水平的監督崩潰
- R embed step_discretize_xgb 使用 XgBoost 離散數值變量
- R embed step_pca_sparse_bayes 稀疏貝葉斯 PCA 信號提取
- R embed step_lencode_mixed 使用貝葉斯似然編碼將監督因子轉換為線性函數
- R embed step_embed 將因子編碼到多列中
- R embed step_woe 證據權重變換
- R embed step_discretize_cart 使用 CART 離散數值變量
- R embed step_feature_hash 通過特征哈希創建虛擬變量
- R embed dictionary 證據權重詞典
- R embed is_tf_available 測試一下tensorflow是否可用
- R embed add_woe 在 DataFrame 中添加 WoE
- R SparkR eq_null_safe用法及代碼示例
- R SparkR except用法及代碼示例
- R SparkR explain用法及代碼示例
- R SparkR exceptAll用法及代碼示例
- R dtrMatrix-class 三角形稠密數值矩陣
- R vcov.gam 從 GAM 擬合中提取參數(估計器)協方差矩陣
- R gam.check 擬合 gam 模型的一些診斷
- R ggplot2 annotation_logticks 注釋:記錄刻度線
- R matrix轉list用法及代碼示例
注:本文由純淨天空篩選整理自Max Kuhn等大神的英文原創作品 Supervised and unsupervised uniform manifold approximation and projection (UMAP)。非經特殊聲明,原始代碼版權歸原作者所有,本譯文未經允許或授權,請勿轉載或複製。
